抗体优化¶
本任务可以帮助您使用强大、专业的 AI 模型,进行抗体评价、筛选和优化。
如您当前有一条先导抗体,您可以用本任务对其进行一键人源化,随后对生成的序列进行虚拟筛选,选出最佳的分子进行湿实验验证。
如您当前有多条先导抗体,您可以使用本任务对它们的人源性、表面电荷、疏水性、等电点、表达量、结构稳定性、PTM 风险等指标进行综合打分,并根据打分结果进行分子的排序、筛选。氨基酸级别的打分高低会在序列和结构上用颜色直观展示出来。
如您需要改造任何已有的抗体,以获得更高的亲和力、稳定性和可开发性,本任务中的抗体突变助手会为您智能推荐突变方案,提示突变方向,助您高效进行抗体改造。
输入¶
要提交抗体优化任务,请打开项目编辑器 并点击左侧边栏中的"New Job(新建任务)"按钮,然后选择"Antibody Design(抗体设计)"任务组中的"Antibody Optimization(抗体优化)"。任务提交表单将在新标签页中打开。
- Sequence type(序列类型): 待优化的抗体类型。可选“VH+VL”(默认)和“VH”。
- Antibodies(抗体): 待优化/评价/筛选的一条或多条抗体。您可以在输入框中输入序列,也可以上传 FASTA 文件 (点击 "
" 按钮)或将其内容粘贴到输入框中。
- Antibody Name(抗体名称): 抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
" 按钮.
- Sequence(序列): 输入每条抗体的 VH/VL 区序列。
(上传序列): 点击 "
" 按钮上传您自己的抗体(一个 .FASTA 文件)。您的抗体必须有两条链,且标签必须具有相同的前缀且分别以 "_H" 和 "_L" 结尾。参见以上示例。
- Antibody Name(抗体名称): 抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
- Job Name(任务名称): 任务的名称。请注意,任务名称必须在项目内唯一。

参数¶
本任务的参数包括:
- Scheme(编号方案): 为抗体每个氨基酸进行编号,使不同抗体的等效氨基酸互相对齐的方案。可选“Kabat”、“IMGT”、“Chothia”或“AHo”。
- CDR def.(CDR 定义): CDR 区域的定义,会影响 CDR 嫁接和一键人源化的结果。可选“Kabat”、“IMGT”、“Chothia”或“North”。这里对比了不同定义方案的区别。当您需要进行一键人源化时,推荐使用“North”。
- Species(物种): 抗体的目标作用物种,会用于检索 VH/VL 胚系基因。可选“human”(默认)、“cat”、“mouse”、“rat”、“rabbit”、“alpaca”、“pig”和“rhesus”。
-
Mutator(突变方案):用于突变起始序列的方案。只有当输入序列只有一条时,才能进行突变。可选的突变方案包括:
突变方案
不进行突变。结果总表中初始的抗体与输入的抗体相同。
将输入抗体的 CDR 区域嫁接到人源胚系基因上的传统人源化技术。结果总表中将为输入序列创建一条人源化后的序列。下面是该突变方案的参数:
- VH germline(VH 胚系基因): 用于 CDR 嫁接的 VH 胚系基因。默认为“Auto”,即自动选择指定物种中与输入序列最相近的 VH 胚系基因。
- VL germline(VL 胚系基因): 用于 CDR 嫁接的 VL 胚系基因。默认为“Auto”,即自动选择指定物种中与输入序列最相近的 VL 胚系基因。
- Keep Vernier(保留 Vernier 区氨基酸): 是否保留 Vernier 区氨基酸,以维持亲和力。
- Keep VH-VL interface(保留 VH-VL 界面氨基酸): 是否保留 VH-VL 界面氨基酸,以维持抗体稳定性。仅适用于“VH+VL”类型的抗体。
- Keep Nb KeyRes(保留纳米抗体关键氨基酸): 是否保留纳米抗体关键氨基酸,以维持纳米抗体可溶性和稳定性。仅适用于“VH”类型的抗体。
使用百奥几何研发的人源化算法将输入的抗体进行人源化。结果总表中将为输入序列创建一条人源化后的序列。下面是该突变方案的参数:
- # Iterations:GeoHumAb 算法的人源化迭代次数。默认值为 1。值越大,序列人源性越高,但序列保守性越低。
- Keep CDRs(保留 CDR 区氨基酸): 是否保留 CDR 区氨基酸,以维持亲和力。
- Keep Vernier(保留 Vernier 区氨基酸): 是否保留 Vernier 区氨基酸,以维持亲和力。
- Keep VH-VL interface(保留 VH-VL 界面氨基酸): 是否保留 VH-VL 界面氨基酸,以维持抗体稳定性。仅适用于“VH+VL”类型的抗体。
- Keep Nb KeyRes(保留纳米抗体关键氨基酸): 是否保留纳米抗体关键氨基酸,以维持纳米抗体可溶性和稳定性。仅适用于“VH”类型的抗体。
仅适用于“Species(物种)”为“human”的任务。
使用百奥几何研发的一键人源化算法,一键进行轻重链配对、CDR 嫁接、回复突变选择,得到多条人源化后的序列,供后续计算参考。结果总表中将为输入序列创建多条人源化后的序列。下面是该突变方案的参数:
- VH germline(VH 胚系基因): 用于 CDR 嫁接的 VH 胚系基因。默认为“Auto”,即自动选择与输入序列最相近的人类 VH 胚系基因。
- VL germline(VL 胚系基因): 用于 CDR 嫁接的 VL 胚系基因。默认为“Auto”,即自动选择与输入序列最相近的人类 VL 胚系基因。
仅适用于“Species(物种)”为“human”的任务。
结果¶
在文件与任务管理器面板中点击您之前创建过的任务名,即可查看任务结果。

本任务的结果会在三个页面中展示:
- 总结页:列出所有抗体分子的各项性质。您可以在此页添加新的抗体序列以及新的待预测的性质。
- 对比页:对齐您选中的抗体序列,并对比它们的氨基酸级别性质打分。
- 编辑页:在 AI 辅助下优化某条抗体序列,产生各项性质更优的抗体分子。
总结页¶
在结果面板(上图)的 Antibody Properties(抗体性质)表格中,列出了任务中的所有抗体分子的名字(Name)、VH ID、VL ID(如果序列类型为“VH+VL”)及其各项性质的预测值。
这些抗体可能来自任务输入、突变算法生成、或您在编辑页上的手动编辑。表格中列出的性质包括:
结果总表中列出的抗体性质
- Name:抗体的名称。您可以点击文字右侧的“
”按钮重命名抗体。请注意,抗体名称在一个任务的所有抗体中内必须唯一。
- VH ID:抗体的 VH 区序列 ID。您可以点击文字右侧的“
”按钮重命名 VH ID。重命名时,所有 VH 序列相同的序列都将被重命名。
- VL ID:抗体的 VL 区序列 ID。您可以点击文字右侧的“
”按钮重命名 VL ID。重命名时,所有 VL 序列相同的序列都将被重命名。
- Humanness (percentile):抗体的人源性评分及其在抗体库中的百分位。一般认为,百分位低于10%的抗体有较高的免疫原性风险。
- Germline content:抗体中与胚系基因序列相同的氨基酸的占比。一般需要大于80%。
- FR Germline content:抗体 FR 区中与胚系基因序列相同的氨基酸的占比。一般需要大于85%。
- Germline allele:抗体的胚系基因。轻重链基因用“|“分隔,如“IGHV1-2*02 | IGKV4-1*01”。
- Optimal pH:抗体结构稳定性最高的 pH 值。
- pI:根据结构计算的抗体等电点。
- Positive patch (percentile):抗体表面正电荷块的数量及其百分位。百分位超过 90%,表示分子有较高的聚集性风险。
- Negative patch (percentile):抗体表面负电荷块的数量及其百分位。百分位超过 90%,表示分子有较高的聚集性风险。
- Charge symmetry (percentile):抗体 VH 和 VL 的电荷对称性(表面氨基酸净电荷之积)。百分位小于 10%,表示分子有较高的聚集性风险。
- Hydrophobic patch (percentile):抗体表面疏水氨基酸块的数量。百分位小于 10% 或大于 90%,表示分子有较高的聚集性风险。
- Thermostability (percentile):抗体热稳定性。百分位小于 10%,表示分子有较高的热稳定性风险。目前仅支持“VH”类型的抗体(即纳米抗体)。
- Expression (percentile):抗体在 CHO 细胞中的表达量。百分位小于 10%,表示表达量较低。目前仅支持“VH+VL”类型的抗体。
- Purity (percentile):抗体一步 A 柱后的纯度(以SEC-HPLC检测)。百分位小于 10%,表示纯度较低。目前仅支持“VH+VL”类型的抗体。
- #PTMs:抗体翻译后修饰(PTM)、非特异性结合风险位点的数量。
- #PTMs (CDR/Vernier):抗体 CDR 区和 Vernier 区内翻译后修饰(PTM)、非特异性结合风险位点的数量。您可以将鼠标悬浮在数字上,查看关于风险类型和所在位点的详情。
- Energy (Amber14SB):使用 Amber14SB 力场计算的抗体结构能量。
- plDDT:模型预测的、整个抗体的 lDDT(局部距离差异测试)值,在 0-100 之间。值越大,抗体结构预测的置信度越高。与结构稳定性有一定正相关性。
- CDR plDDT:模型预测的、抗体 CDR 区域的 lDDT 值,在 0-100 之间。值越大,抗体 CDR 区域的结构预测的置信度越高。
- wpTm:模型预测的加权 TM 值,在 0-1 之间。值越大,抗体 VH-VL 复合物的结构预测的置信度越高。与轻重链配对倾向有一定正相关性。
- RMSD to {{ref}}:抗体结构与参考结构之间的 RMSD。
- CDR RMSD to {{ref}}:抗体 CDR 结构与参考 CDR 结构之间的 RMSD。越低说明突变体的 CDR 结构与参考结构越相似,即亲和力维持得可能会更好。
- ΔFitness (heavy/light) to {{ref}}: 突变体重链/轻链与参考序列相比适应性打分的变化。越高说明突变体越有可能具有更好的适应性(可开发性/亲和力)。
以上列默认不会出现。您点击表格上方的“”并指定参考序列、列名中{{ref}}占位符的内容后,这些列才会出现。
每个性质列都包含两个操作按钮:
排序:点击上下两个三角可以对当前列按数值大小进行升序、降序排序。
过滤:点击后可以对当前列数值进行过滤。
表格上方有五个操作按钮:
| 图标 | 操作 | 描述 |
|---|---|---|
| 自定义列 | 在弹出的窗口中自定义表格中列出的性质及其顺序。当您只想观察对比某几列时,可以点击这个按钮隐藏掉其他列。 | |
| 批量操作 | 点击后可以对表格中您选中的的多个抗体进行批量操作。目前支持的操作包括:
|
|
| 增加突变体打分列 | 在表格中增加若干列,展示抗体作为某个参考抗体的突变体的结构相似性/适应性变化打分。 | |
| 添加序列 | 在弹窗中输入抗体序列,并将其添加到本任务的抗体列表中。 | |
| 对比序列 | 对齐并对比您选中的抗体序列。点击后将跳转到对比页,展示各抗体的分子级别打分和氨基酸级别打分。 |
对比页¶
本页(标题为“Antibody Property Comparison”)会对您选中的抗体进行序列对齐,并对比其分子打分和氨基酸打分。

点击标题右侧的括号中的任务名可返回总结页。

本页由“Antibody Properties(抗体性质列表)”和“Per-residue scores(氨基酸级别打分)”两部分组成。
Antibody Properties(抗体性质列表)¶
在“Antibody Properties”一节中,以表格形式列出了抗体的各项性质。和总结页中一样,您可以点击按钮自定义表格中展示的性质及其顺序,也可以点击
按钮对选中的抗体进行批量操作,如复制 FASTA 序列、导出 CSV 表格。
Per-residue scores(氨基酸级别打分)¶
在“Per-residue scores”一节中,对齐展示了您选中的抗体序列及模型对每个氨基酸的多维度评分。您可以在选项卡中切换用于打分的模型。
每个氨基酸的背景色代表了当前模型对该残基的评分。具体规则如下:
氨基酸打分的上色方案
展示氨基酸类型。
展示氨基酸在人体中引起免疫原性的风险。无风险则为白色;红色越深,免疫原性风险越高。
具体地说,如果一个 9-肽片段在抗体数据库中在低于 10% 的人类中出现,则该片段被认为是高风险肽段。氨基酸属于越多高风险肽段,其红色越深。
如果您在编辑页中打开本打分模型,当前序列中有免疫原性风险的氨基酸(在当前序列所在的胚系基因家族,如IGHV1中,当前氨基酸出现的频率低于1%)会被标上红色三角形。如果在编辑前当前位点有免疫原性风险,但编辑后已被消除,则位点会被标上绿色三角形
。
展示基于结构计算的氨基酸侧链的 pKa。蓝色越深,pKa 越高(酸性越弱);红色越深,pKa 越低(酸性越强)。
展示基于结构计算的氨基酸的表面净电荷。正值为蓝色,负值为红色。
展示基于结构计算的抗体 CDR 区(IMGT定义)及邻域的表面净电荷块大小。默认为白色;同性净电荷块越大,由电荷导致的聚集性风险越高,红色越深。
展示氨基酸的疏水性。黄色越深,疏水性越高;青色越深,疏水性越低。
展示基于结构计算的抗体 CDR 区(IMGT定义)及邻域的疏水块大小。默认为白色;疏水块越大,由疏水导致的聚集性风险越高,红色越深。
展示基于序列和结构计算的抗体翻译后修饰(PTM)和非特异结合位点。默认为白色;PTM/非特异结合风险越高,红色越深。
除背景色外,本节还存在下列形式的序列标注:
- CDR 区氨基酸用深灰色下划线标出,Vernier 区、VH-VL界面区氨基酸用浅灰色标出。这些氨基酸在突变时可能影响亲和力或稳定性,请谨慎修改。
- 如果您在设置中开启了“Show solvent accessibility(展示氨基酸溶剂可及性)”,氨基酸的溶剂可及性会用字符颜色深浅和字重表示。黑色、加粗的氨基酸暴露在溶剂中,而灰色、加细的氨基酸则相反。
- 仅有参考序列会被“完全展示”,即每个氨基酸都用大写字母表示。其余序列中出现"·"的位置,其氨基酸与参考序列相同。出现大写字母的位置,表示其氨基酸与参考序列不同。您可以点击序列右侧的标识符(抗体名称或 VH/VL ID)来切换参考序列。
- 某些序列的某些位置上可能没有氨基酸。在这种情况下,该位置会显示为“-”(横杠)。
- 鼠标悬浮在某个氨基酸上时,会出现一个浮层展示该氨基酸的详细信息,包括位置编号、所属区域、打分数值等,例如"H113 (IMGT) | CDR3 | score: 0.889"。如果打分模型是 PTM,且该氨基酸有序列风险,则浮层中还会展示序列风险的详情。详情包括:风险类型、freq(当前位置出现当前风险的频率)、rsa(当前氨基酸的溶剂可及性)。

在表格上方有四个按钮,用于执行不同的操作:
| 图标 | 操作 | 描述 |
|---|---|---|
| 添加序列 | 添加新的序列到本对比页中。新的序列可以来自当前任务,也可能来自胚系基因搜索。 | |
| 显示所有/唯一序列 | 在显示所有选中的序列(序列以抗体名区分)和仅显示唯一序列(序列以 VH/VL ID 区分)之间切换。 | |
| 切换轻重链 | 切换显示重链或轻链。 | |
| 设置 | 打开设置浮窗,您可以修改以下配置项:
|
编辑页¶
本页(标题为“Editing {antibody_name}”)允许您在 AI 辅助下编辑某条抗体序列,产生各项性质更优的抗体分子,并将其保存到结果总表中。

点击标题右侧的括号中的任务名可返回总结页。

本页包括如下区域:
- Antibody Properties(抗体性质列表):展示编辑前和当前抗体的各项性质
- Per-residue scores(氨基酸级别打分):对齐展示了编辑前和当前的抗体序列及模型对每个氨基酸的多维度评分。
- Mutation advisor(突变助手):为当前抗体序列提供突变建议
- Structure viewer(结构查看器):显示当前抗体的结构
前两个区域和对比页几乎完全相同,主要区别在于:
- 您可以点击“Save”按钮将当前序列保存到结果总表中。保存后,当前序列会作为参考序列,继续编辑页中显示。
- 您可以点击
按钮打开设置浮窗,在“Mutation advisor”配置项中选择您的突变助手。
- 氨基酸的详细信息浮层中,会展示突变助手的相关结果。

突变助手¶
突变助手为您改造抗体序列提供建议。您可以点击按钮打开设置浮窗,在“Mutation advisor”配置项中选择您的突变助手。可选的选项包括:
突变助手设置项

AI 助手会根据您当前的抗体序列,为每个位点给出至多 5 个突变建议(当前氨基酸也会参与排名)。每个位置所有氨基酸的打分之和为 1。氨基酸背景的紫色越深,代表模型认为其在该位置的倾向性越高,如下图。 为了使突变建议更清晰、易读,突变建议中当前序列中的氨基酸残基以点号("·")表示,置信度低于 1% 的氨基酸将被省略。 可选的助手模型(Copilot model)有:
- SaProt:能够同时建模序列和结构的蛋白语言模型。推荐您用此模型进行抗体优化。
- ESM1v-650M:专门用于预测变异影响的语言模型。
- ESM2-3B:更新、更强大的通用蛋白质语言模型。
- GeoHumAb:我们的专有算法,用于抗体的人源化。

展示与当前序列最接近的五条胚系基因序列。其中,带淡蓝色背景的氨基酸与当前序列相同,如下图。您可采用将氨基酸突变到胚系基因序列中对应位置的氨基酸的方式,降低当前序列的免疫原性。
您可在设置中自定义胚系基因的来源物种(Species),可选项包括:Human(人)、Cat(猫)、Mouse(小鼠)、Rat(大鼠)、Rabbit(兔)、Alpaca(羊驼)、Pig(猪)、Rhesus(猕猴)。默认值为 Human。

当您将鼠标悬停在突变建议上时,会出现一个浮层展示当前氨基酸的详细信息,内容包括:
- Position(位置):您悬停的氨基酸所在位置的编号。格式为“{链类型}{残基编号}{插入代码}”,例如“H52A”。
- Region(区域):该位置所属的抗体区段,值可能为 FR1-4 或 CDR1-3。Vernier 区、VH-VL 界面区、纳米抗体关键残基也会被标出。这些氨基酸在突变时可能影响亲和力或稳定性,请谨慎修改。
- Advisor confidence(突变建议的置信度):排名前 10 的突变建议及其置信度,鼠标悬停处的氨基酸残基以蓝色高亮显示。置信度越高,代表该突变越有可能改善抗体性质(人源性/适应性)。
- Risk detail:如果当前打分模型为 PTM,且氨基酸有风险,浮层中还会展示风险详情。详情包括:风险类型、freq(当前位置出现当前风险的频率)、rsa(当前氨基酸的溶剂可及性)
下面列出了编辑当前序列的方法
编辑当前序列的方法
将鼠标悬浮在“Current (mt)”右边的图标上,选择“Manually edit sequence”进入序列编辑模式。此时 Current (mt) 序列的下方会出现一条可编辑的序列,您可以:
- 点击需要编辑的位置,用键盘输入您想突变的氨基酸。按下 Tab、Shift+Tab 键可以在各个氨基酸之间左右移动。
- 点击最左边的位置,按 Ctrl+V 粘贴突变后的序列。
- 单击氨基酸级别打分或突变建议中的任意氨基酸,将当前位点的氨基酸突变为该氨基酸。
当您突变完毕后,点击可编辑序列右方的 OK,即可更新当前序列。

如您想将当前序列人源化,您可以将鼠标悬浮在“Current (mt)”右边的图标上,选择“Humanize sequence”进入人源化弹窗。设置人源化相关参数后点击“Confirm”即可触发人源化计算,计算完毕后 Current (mt) 序列的下方会出现一条可编辑的序列,您可以和自定义突变时一样,继续编辑该序列。编辑完成后,点击可编辑序列右方的 OK,即可更新当前序列。
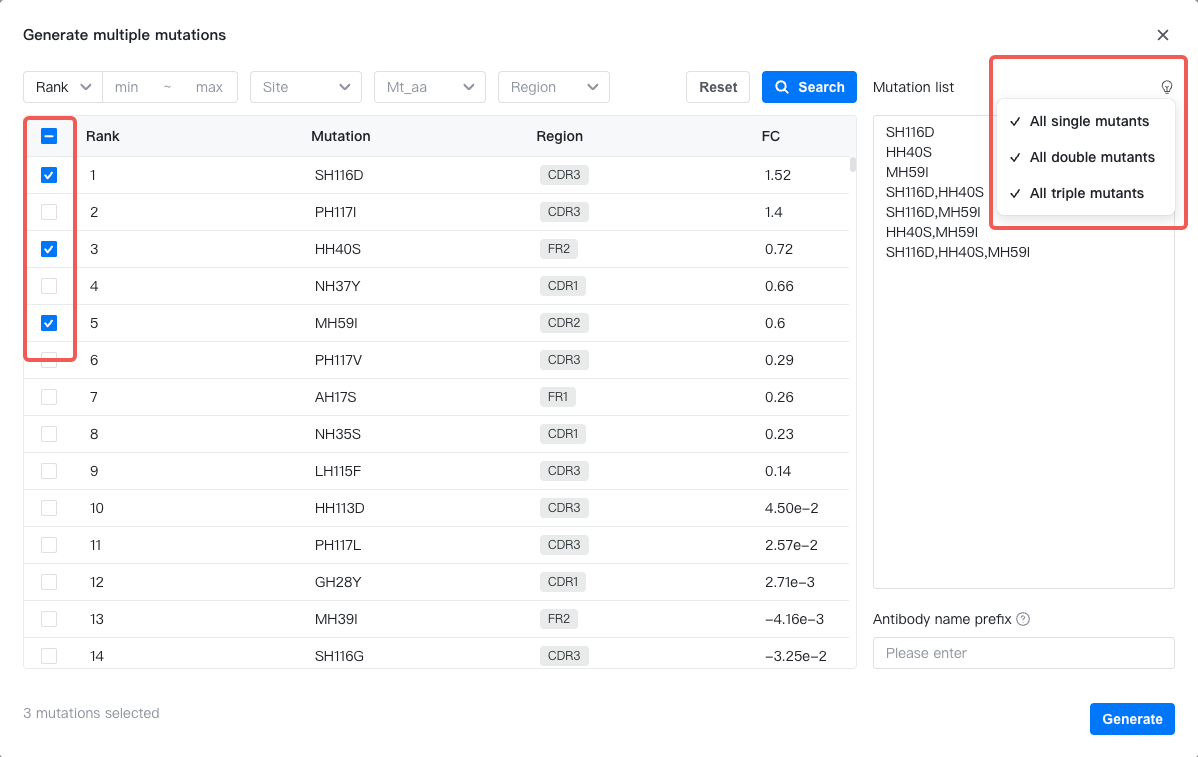
将鼠标悬浮在“Current (mt)”右边的图标上,选择“Generate multiple mutants”进入突变体生成弹窗。
弹窗左侧的表格中列出了AI助手给出的突变建议。您可以使用表格的筛选功能筛选您希望列出的突变。您选择想要应用的点突变后,可点击面板右侧的按钮生成这些点突变的所有单突变、双突变和三突变体。
您也可以手动输入突变列表。
当您准备好突变列表后,在“Antibody name prefix”输入框中输入这些新抗体分子名称的前缀,然后点击“Generate”就可以将它们加入结果总表中了。
结构查看器¶
结构查看器显示了当前抗体的结构。
结构中的氨基酸残基会按照与序列相同的方案进行着色,以反映当前模型对每个氨基酸的评分。
致谢¶
本任务由以下模型提供支持:
- 百奥几何自研的抗体结构预测模型,
- 百奥几何自研的抗体人源化和人源性预测模型,
- 百奥几何自研的表达量/聚集性/热稳定性预测模型,
- 西湖大学提出的 SaProt 模型,用于蛋白质序列的建模和优化,
- Meta AI 的 ESM系列模型,用于蛋白质序列的建模和优化,
- 牛津大学提出、百奥几何改良的抗体成药性分析(TAP)模型,可预测抗体侧链 pKa、表面电荷、疏水性、CDR 长度等性质,并与临床阶段抗体进行比较。
- 百奥几何自研的序列风险分析模型,用于预测翻译后修饰(PTM)、非特异结合等序列风险的分析预测。
