从头抗体设计¶
从头抗体设计是一项针对给定抗原/表位从头设计结合抗体的任务。它对于针对"难成药"抗原的抗体发现尤其有价值。
挑战¶
从头抗体设计的搜索空间非常庞大,因为我们需要在考虑抗体稳定性和成药性的同时,确定抗体结合位点中 30 多个残基的氨基酸类型和位置。传统方法难以在效率、新颖性和准确性之间取得平衡,而 BioGeometry 的生成式 AI 方法如 GeoFlow 在从头抗体设计中实现了业界领先的 18.7% 平均命中率。
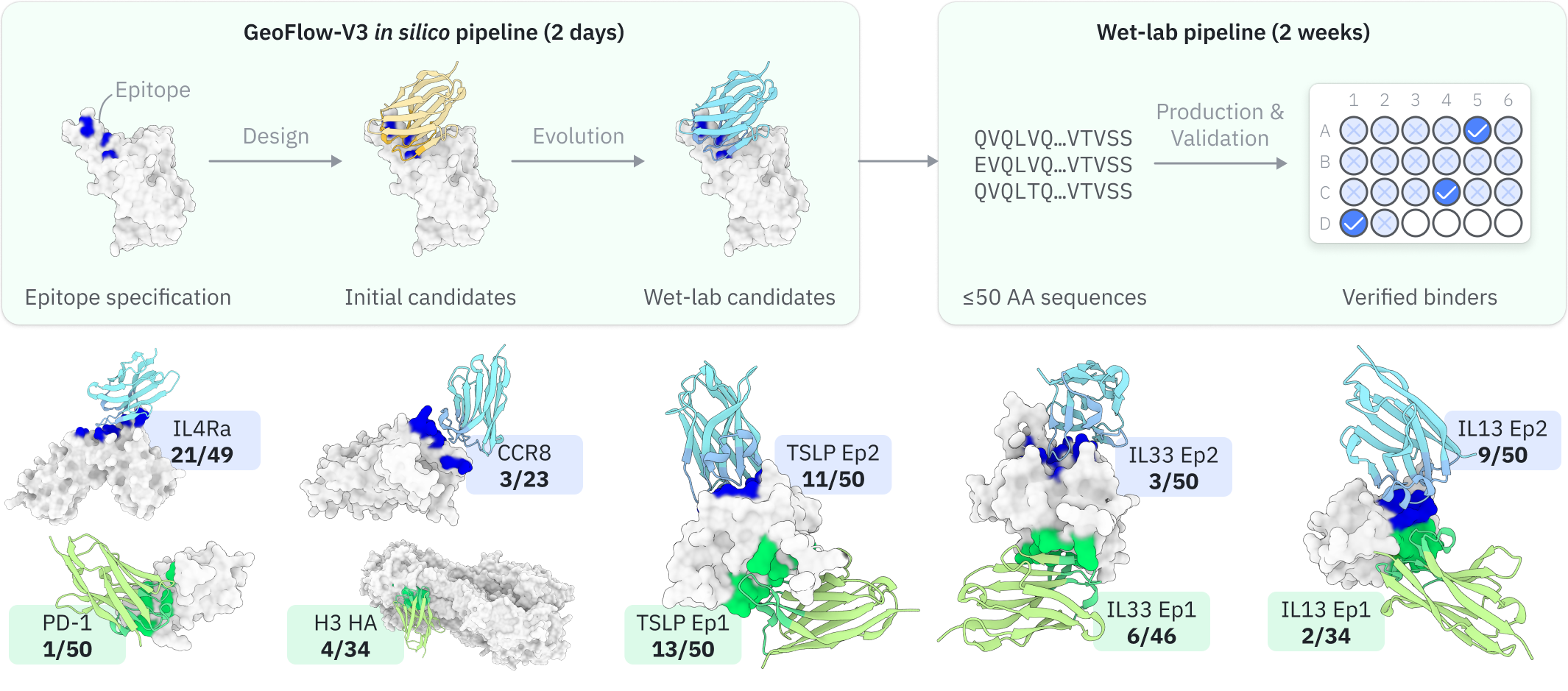
流程概述¶
本节将对 GeoFlow-V3 从头抗体设计流程进行高层次的概述,以帮助您实现更好的设计结果。
- 从头设计: GeoFlow 以抗原和部分抗体序列作为输入,针对指定的目标表位设计抗体的初始序列和结构。
- 虚拟筛选: GeoFlow 以完整的抗原:抗体序列作为输入,输出置信度指标和预测结构,然后用于计算自洽性指标和抗体成药性指标。这些指标用于筛选出顶级候选分子以进行进一步优化。
-
虚拟进化: 如果任务输入中开启了"evolution(进化)",GeoFlow 将对顶级候选分子执行多轮虚拟进化,以进一步提高顶级候选分子的结合率。每轮进化包括对选定抗体区域的部分重新设计和顶级候选分子选择。最终结果是最后一轮进化中的顶级候选分子。
In silico 抗体进化过程示意图。
输入¶
要提交从头抗体设计任务,请打开项目编辑器并点击左侧边栏中的 "New Job(新建任务)" 按钮。然后点击 "Antibody Design(抗体设计)" 任务组下的 "De Novo Antibody Design(从头抗体设计)" 以打开任务提交页面。
-
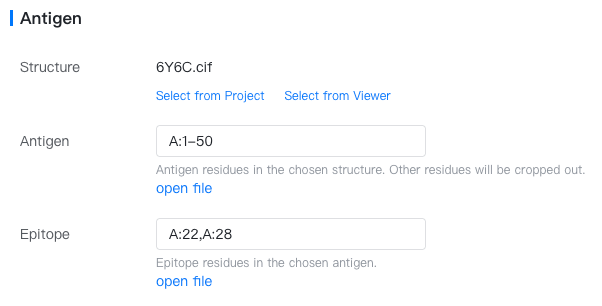
Antigen(抗原): 目标抗原。
-
Structure(结构): 选择包含所需抗原结构的任何 PDB 或 mmCIF 文件。如果该文件已经在结构查看器中打开,请点击 "Select from viewer"。如果该文件在您当前的项目中,请点击 "Select from project"。否则,您可以在文件与任务管理器中上传或从 PDB 等云数据库中导入。
-
Antigen(抗原): 指定抗原残基,例如
A:3-27,B:10-50。请注意,多条抗原链之间的相对位置将保持不变。 -
Epitope(表位): 构成抗原表位(抗体结合位点)的一组残基。输入以逗号分隔的连续序列片段列表,格式为
{chain}:{start_res}-{end_res},例如A:2-10,A:15-30,B:40-100。您还可以使用结构查看器从抗原结构中选择表位,然后点击 "import from selection" 以自动填充此输入框。
表位格式
在 GeoBiologics 中,表位残基列表由一个或多个"位点"和"片段"组成。
- 每个位点表示为
{chain_id}:{res_id},例如H:100。 - 每个片段表示为
{chain_id}:{start_res_id}-{end_res_id}。例如H:100-112。请注意,起始和结束残基 ID 是 label residue IDs 而不是 author residue IDs。
多个位点或片段可以用逗号连接,例如
H:100-110,H:111,H:111A,H:112B,L:50-60,L:92。从选择中导入抗原/表位
点击 "Antigen(抗原)" 或 "Epitope(表位)" 输入框,您将在输入框下方看到 "open file" 或 "import from selection"。
- 如果输入结构文件未在结构查看器中打开,您可以点击 "open file" 打开它。
- 如果输入结构文件已在结构查看器中打开,您可以进入选择模式并在结构上选择突变位点。然后点击 "import from selection" 以自动填充此输入框。
- 您还可以运行界面可视化任务,该任务将在结构查看器中创建界面"组件"。然后您可以选择抗原与抗体的界面并点击 "import from selection" 以自动填充此输入框。
-
-
Antibody(抗体): 具有待设计 CDR 区的抗体。
- Sequence type(序列类型): VH+VL(IgG)或 VH(纳米抗体)。
- Antibody Name(抗体名称): 抗体的名称。默认为 "Antibody 1"。要更改它,请将鼠标悬停在抗体名称上方,然后点击 "
" 按钮。
- Sequence(序列): 指定抗体每个区域(FR1, CDR1, ..., FR4)的模板序列。待设计的 CDR 区残基需要指定为 "X"。如您要设计可变长度区域,请输入 "[X, {min_length}-{max_length}]",例如 [X, 12-18]。FR 区残基不能被设计。
CDR 模板示例
[X, 12-12]: 设计整个区域(12 个残基)。[X, 10-18]: 设计整个区域(10-18 个残基)。AR[X,3-6]A[X,3-3]: 部分重新设计。固定 3 个残基,在第一个片段中设计 3-6 个残基,在第二个片段中设计 3 个残基。AR[X,3-5]CDXXTX: 部分重新设计。固定 5 个残基,在第一个片段(AR之后)中设计 3-5 个残基,在第二个片段(CD之后)中设计 2 个残基,在第三个片段(T之后)中设计 1 个残基。
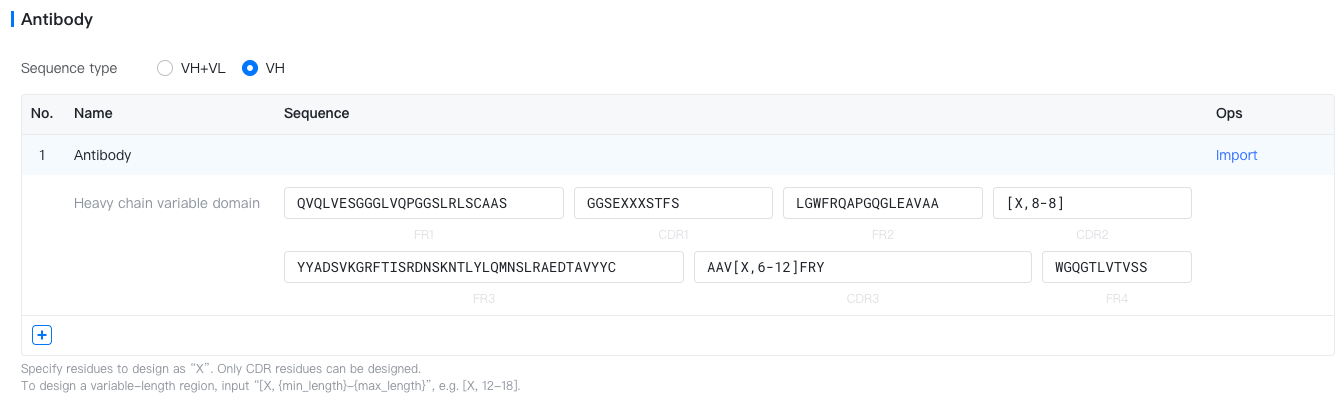
- Import(导入): 点击 "Import" 文本以从已验证的抗体(例如 Trastuzumab)或您自定义的抗体序列出发,填写抗体序列模板。
-
Job Name(任务名称): 任务的名称。请注意,任务名称在项目中必须是唯一的。
模型 & 参数¶
我们用于抗体设计的专有生成模型 GeoFlowV3 可用于此任务。该模型的参数如下。
- # Structure(设计结构数): 生成的结构数量(默认为 10000)。如果您的输入包含多条抗体模板,每个生成的结构会随机选择一个抗体模板。
- # Seq/Struct(每个结构的序列数): 每个有效结构生成的序列数量(默认为 40)。
- Experiment budget(实验预算): 在代表性结构表中返回的设计数量(默认为 40)。如果您计划对设计结果进行手动结构检查,请指定比实际实验预算大 1-2 倍的数字。
参数选择提示
在实际项目中,建议您首先尝试运行 ~1000 个结构 × 4 个序列/结构 来验证表位,然后根据第一次运行中的虚拟筛选通过率调整参数。如果通过率较低,请尝试增加设计结构和序列的数量以获得足够的高排名候选分子。
结果¶
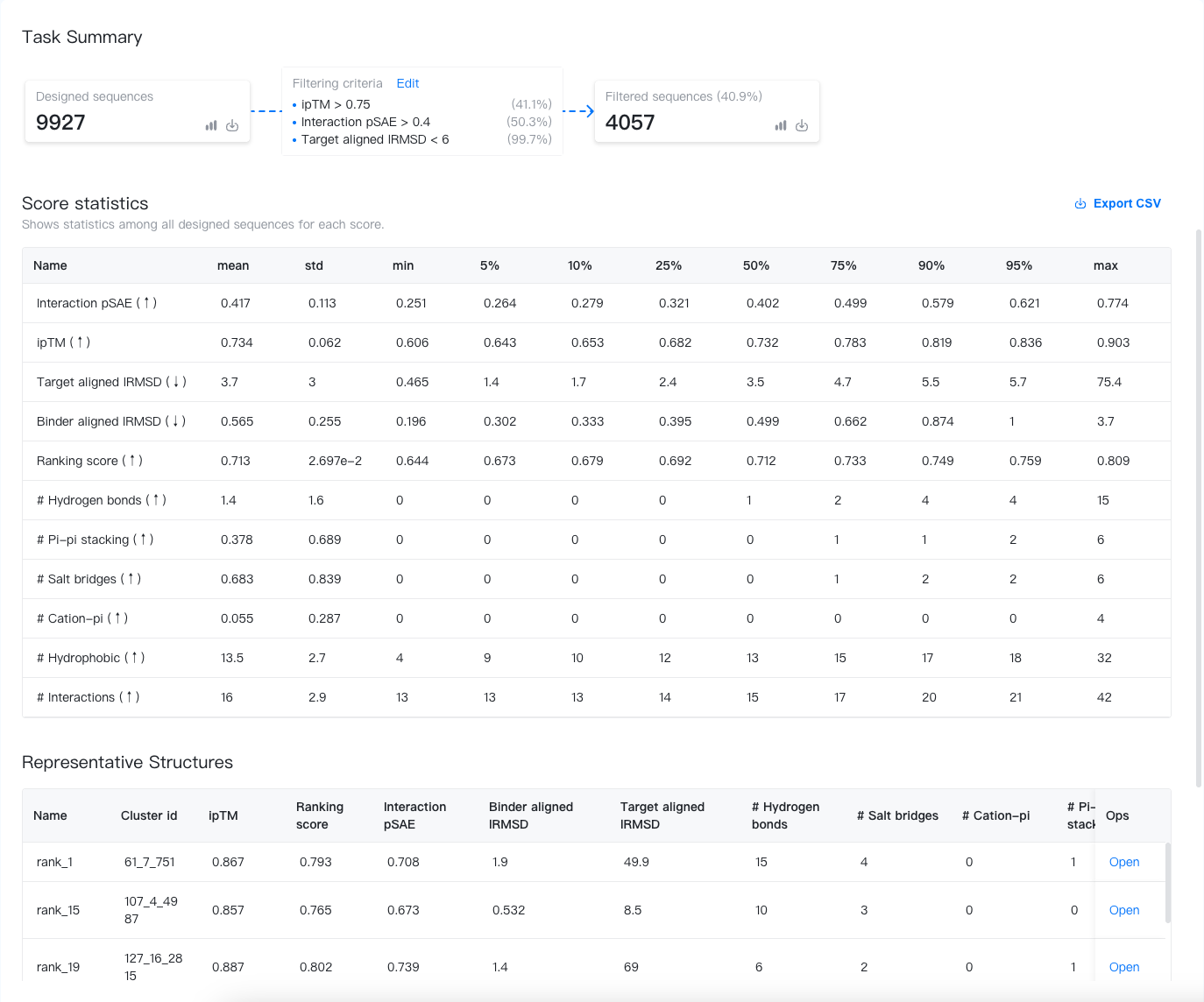
任务小结¶
任务小结展示了有关设计序列、筛选条件和筛选后序列的关键信息。让我们在此详细说明:
- Designed sequences(设计后的序列): 粗体数字显示了设计序列的总数。
: 点击预览前 500 个设计的序列及其分数。
: 鼠标悬停以下载(1)设计区域的序列及所有相应分数的 .csv 文件;(2)完整设计序列的 .FASTA 文件;(3)所有设计序列的 .tar.gz 文件。
- Filtered sequences(筛选后的序列): 粗体数字显示了筛选后序列的总数。百分比是通过虚拟筛选过滤器的设计序列的比率。
: 点击预览前 500 个筛选后的序列及其分数。
: 点击下载(1)筛选区域的序列及所有相应分数的 .csv 文件;(2)完整筛选序列的 .FASTA 文件。
- Filtering criteria(筛选条件): 虚拟筛选中使用的筛选条件。每个条件右侧都有一个百分比,表示通过该条件的序列的比率。您可以通过点击 "Edit" 来自定义筛选条件。筛选后的序列将相应更新。
虚拟筛选中计算的分数
虚拟筛选过程基于我们专有的 GeoFlow 模型,该模型通过计算以下分数来检查设计序列和结构之间的一致性:
- ipTM: 抗体:抗原复合物的预测界面 TM (Template modeling) 分数。分数越高表示界面区域的置信度越高。
- Ranking score(排名打分): AlphaFold3 排名中使用的分数。它基本上是通过 \(\mathrm{wpTM} = 0.8 \times \mathrm{ipTM} + 0.2 \times \mathrm{pTM}\) 计算的预测加权 TM 分数,加上对无序区域和冲突的一些惩罚项。
- Interaction pSAE(相互作用 pSAE): 在抗体-抗原残基对上平均的预测对齐误差分数。分数越高表示预测的抗体-抗原相互作用置信度越高。
- Target-aligned lRMSD(抗原对齐后的抗体 RMSD): 在对齐预测和设计结构中的靶点(抗原)后,结构中抗体的均方根偏差(RMSD)。6Å 以内的分数表示良好的表位一致性。如果预测的表位(抗体结合位点)与设计结构相距较远,这项分数可能会很高。
- Binder-aligned lRMSD(抗体 RMSD): 将预测的抗体与设计的抗体对齐时,抗体结构的均方根偏差(RMSD)。3Å 以内的分数表示良好的抗体结构一致性。这项分数一般都比较低。
- # Hydrogen bonds(氢键数): 抗体和抗原之间的氢键数量。
- # Pi-pi stacking(Pi-pi 堆积数): 抗体和抗原之间的 π-π 堆积相互作用数量。
- # Salt bridges(盐桥数): 抗体和抗原之间的盐桥数量。
- # Cation-pi(阳离子-pi 数): 抗体和抗原之间的阳离子-π 相互作用数量。
- # Hydrophobic(疏水相互作用数): 抗体和抗原之间的疏水相互作用数量。
- # Interactions(相互作用数): 抗体和抗原之间的非共价相互作用总数(以上各项互作数目之和)。
分数统计¶
本节显示了所有设计序列的分数统计信息。显示的统计信息包括均值、标准差、最小值和最大值,以及 5%、10%、25%、50%、75%、90% 和 95% 百分位数。您可以使用这些统计信息来了解分数的分布并设置筛选条件。
代表性结构¶
由于返回所有结构会使您的项目变得混乱,我们仅显示前 k 个(k = Experiment budget,即)结构及其相应的分数。您可以点击 "open" 在结构查看器中打开这些结构。

