人源性预测¶
人源性预测是预测输入抗体序列与人源抗体序列的相似程度的任务。
功能亮点¶
- 指标精确:基于 OAS(Observed Antibody Space)上人源抗体序列大数据,人源性预测高效、准确。
- 界面美观:主要结果清晰易读,种系序列可直观对比,风险位点一目了然。
- 批量预测:支持一个任务提交多条序列到云端批量预测,效率更高、成本更低。
输入¶
要提交一个人源性预测任务, 请打开项目编辑器并从“Characterization”(性质预测)下拉菜单中选择“Humanness Prediction”(人源性预测)。
- Antibodies(抗体):一个用 FASTA 格式表示的抗体序列的列表。您可以在输入框中输入序列,也可以上传 FASTA 文件 (点击 "
" 按钮)。 每个抗体将独立进行预测。
- Chain type(链类型):您可以在 Antibodies 标题的右侧选择抗体的链类型,选项有是“VH”、“VL”或“VH+VL”(默认)。如果您想预测纳米抗体的人源性,请选择“VH”。
- Antibody Name(抗体名称):抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
" 按钮。
- Sequence(序列):抗体的序列。每条链的序列长度不得超过 200 个氨基酸。
(上传序列): 点击 "
" 按钮并选择一个 .FASTA 文件上传您自己的抗体。当 Chain type(链类型)为“VH+VL”时,每个抗体必须有两条链,FASTA 标签具有相同的前缀且分别以 "_H" 和 "_L" 结尾。参见以上示例。
- Job Name(任务名称):任务的名称。请注意,任务名称必须在项目内唯一。

模型 & 参数¶
您可使用我们自研的人源性预测模型 GeoHumAb 来运行本任务。该模型的参数如下。
- scheme(编号方案):抗体编号方案。可选“Kabat”、“IMGT”、“Chothia”或“AHo”。
- CDR def.(CDR 定义):用于定义 CDR 区域的编号方案。可选“Kabat”、“IMGT”、“Chothia”、“North”。默认情况下该参数的值与 scheme(编号方案)相同。当
scheme = AHo时,此参数为必填项。
结果¶
结果小结¶
结果小结存储在 CSV 文件中,可以通过点击结果表格右上角的""按钮下载。
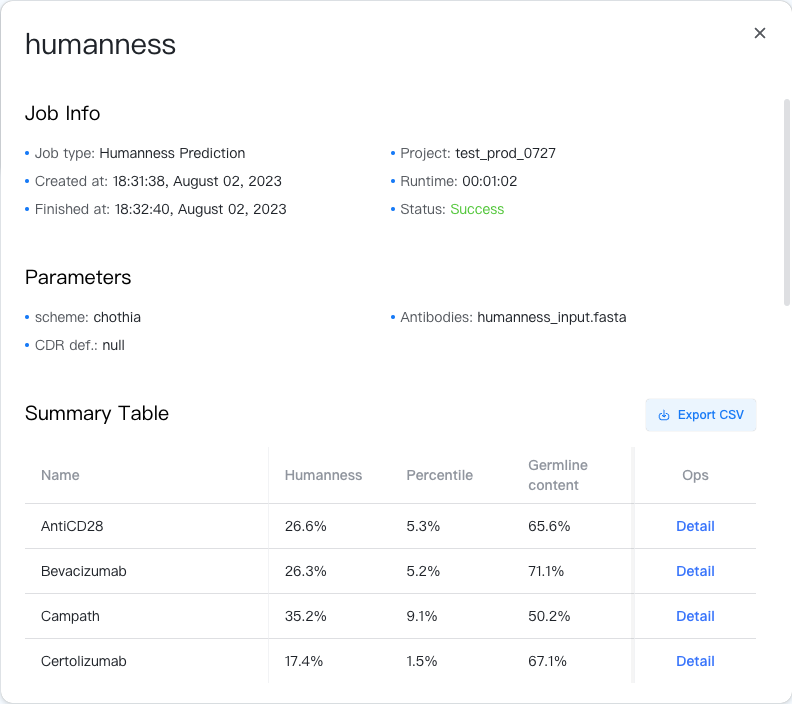
结果表格包含以下列:
- Name(名称):抗体名称,与输入中 FASTA 标签的前缀相同。
- Humanness(人源性):抗体的人源性评分,由抗体中的 9-肽片段在 OAS 数据库人源实验中的平均出现频率决定。得分越高,抗体人源性越好。
- Percentile(百分位):人源性评分在 OAS 数据库中所有抗体中的百分位。得分越高,抗体人源性越好。
- Germline content(种系成分):抗体的种系成分,即抗体与最近的人源轻重链种系序列的序列相似性。
在最右边的列中,您可以点击"Detail"(细节)按钮查看某个抗体人源性的详细结果。
结果详情¶
结果详情页分为 3 个区域:Summary(结果小结)、Germline Comparison(种系比较)和Detailed View(详细视图)。如果输入的链类型为“VH+VL”,种系比较和详细视图区域将分别显示重链和轻链的结果。您可以使用小结部分下方的""链切换器在两条链之间切换。
结果详情的原始数据存储在一个 CSV 文件中。您可以通过点击详情视图区域右上角的""按钮下载。
结果小结¶
结果小结区域显示了任务所用的模型和用于计算人源性的编号方案。它还显示了抗体的人源性评分、百分位和种系成分(如果抗体包含轻重链,显示的值为重链和轻链的平均值)。
序列查看器¶
序列查看器中,对齐显示了抗体链及与其最接近的五条人源种系序列。种系序列的名称显示在右侧,与目标序列相同的种系残基以蓝色高亮显示。
目标序列中种系频率低于 1% 的氨基酸以红色三角形标记,处于高风险肽段中的氨基酸以红色高亮显示。氨基酸属于越多高风险肽段,其红色越深。
CDR 区域的氨基酸残基以深灰色下划线进行了标记。该区域由于 VD(J) 重排和体细胞超突变,可变性较高,因而红色通常较深,但不代表该区域一定存在免疫原性风险。
如果您使用 Kabat 进行 CDR 定义,则 Vernier 区域的氨基酸残基会以浅灰色下划线进行标记。
名词解释
- 种系频率:OAS 数据库相应人源种系中给定位置的氨基酸频率。频率越高,该氨基酸越可能是种系中的氨基酸。
- 高风险肽段:如果一个 9-肽片段在 OAS 数据库人源实验中出现的频率低于 10%,则该片段被认为是高风险肽段。
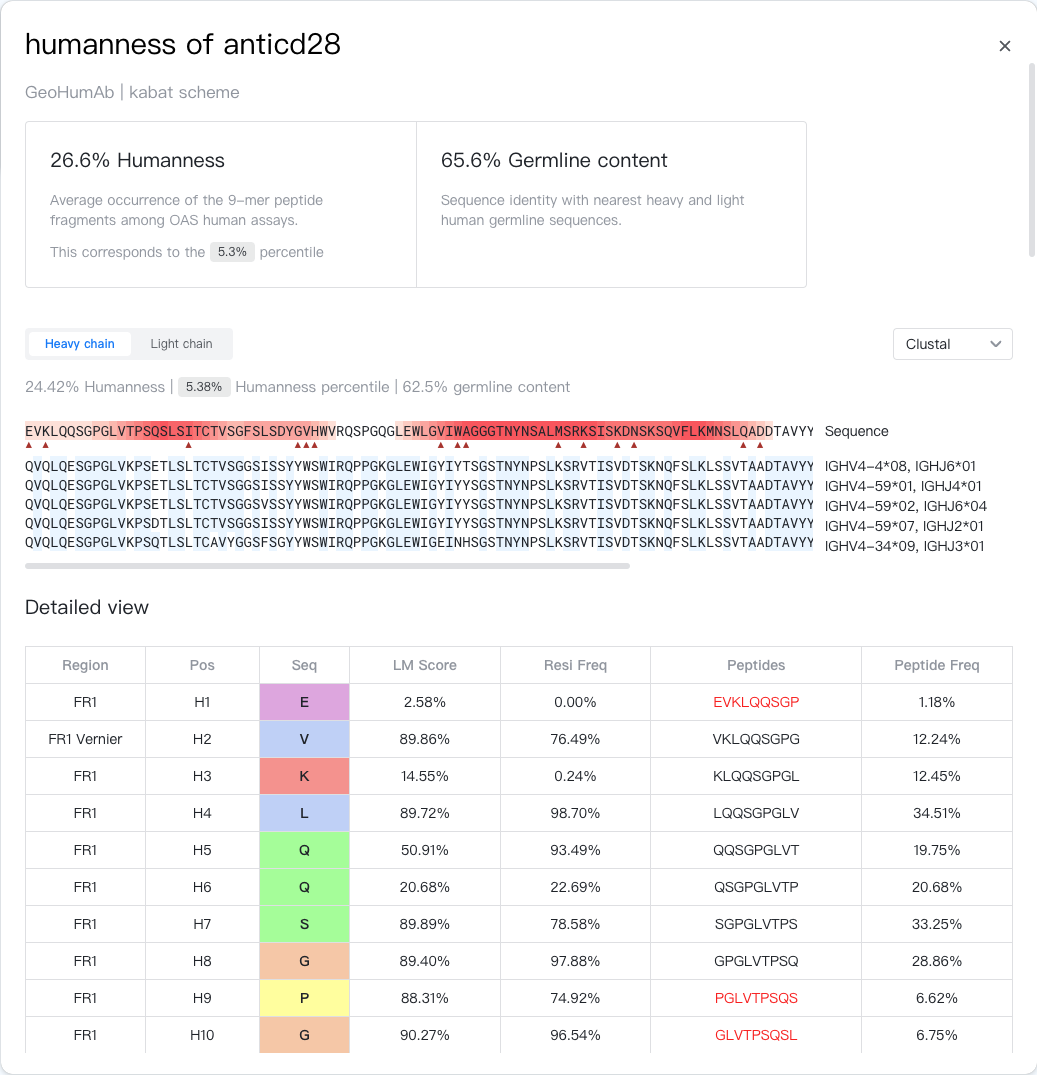
详情视图¶
详情视图区域以表格形式显示抗体链中每个位置的详细指标。表格包含以下列:
- Region(区域):该位置所属的抗体区域,值可能为 FR1-4 或 CDR1-3。区域的计算方法由 CDR 定义决定。如果 CDR 定义设置为 Kabat,则还会标出 Vernier 区域。
- Pos(位置):按照指定方案编号后的抗体链中的位置编号,格式为“{链类型}{氨基酸编号}{插入码}”,例如“H100A”。
- Seq(序列):抗体链上该位置的氨基酸类型。氨基酸按照指定的颜色方案进行着色。您可以使用右上角的"
"颜色方案选择器更改颜色方案。
- LM Score(语言模型分数):该位点的语言模型分数。分数越高,语言模型认为该氨基酸在当前上下文中出现的概率最高。
- Resi Freq(种系频率):OAS 数据库相应人源种系中给定位置的氨基酸频率。频率越高,该氨基酸越可能是种系中的氨基酸。
- Peptides(肽段):以该氨基酸为起始氨基酸的 9-肽片段。如果相应的“Peptide Freq”低于 10%,则该片段以红色显示,被认为是高风险肽段。
- Peptide Freq(肽段频率):该 9-肽片段在 OAS 人源实验中出现的频率。