抗体优化¶
使用强大的专业 AI 助手评估并优化您的先导抗体。供您使用的 AI 助手有:
- 评分助手(scorer):根据多种性质(如人源性、酸碱性和疏水性)对抗体中的每个氨基酸进行评分的 AI。
- 自动突变助手(mutator):将输入的抗体序列突变为初始输出的 AI。
- 突变建议助手(advisor):为您改造抗体提供突变建议的 AI。
- 结构预测助手(structure modeler):精准预测抗体的结构,以便您进行基于结构的可视化分析。
AI 助手由以下模型提供支持:
- 百奥几何自研的抗体结构预测模型,
- 百奥几何自研的抗体人源化和人源性预测模型,
- Meta AI 的ESM系列模型,用于蛋白质序列的建模和优化,
- 侧链 pKa 和疏水性预测模型,
- 序列风险分析模型,用于预测翻译后修饰(PTM)等序列风险的分析预测。
输入¶
要提交抗体优化任务,请打开项目编辑器 并从"优化"下拉菜单中选择"抗体优化"。
- Antibody(抗体): 待优化的抗体。您可以在输入框中输入序列,也可以上传 FASTA 文件 (点击 "
" 按钮)。
- Antibody Name(抗体名称): 抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
" 按钮.
- Sequence(序列): 抗体的序列。每个链的长度不得超过 200 个氨基酸。
(上传序列): 点击 "
" 按钮上传您自己的抗体(一个 .FASTA 文件)。您的抗体必须有两条链,且标签必须具有相同的前缀且分别以 "_H" 和 "_L" 结尾。参见以上示例。
- Antibody Name(抗体名称): 抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
- Job Name(任务名称): 任务的名称。请注意,任务名称必须在项目内唯一。

模型和参数¶
下面是抗体优化中可用的模型和参数的详细信息。
- 通用参数
- Scheme(编号方案): 抗体编号方案。可选“Kabat”、“IMGT”、“Chothia”或“AHo”。
- CDR def.(CDR 定义): 用于定义 CDR 区域的编号方案。可选“Kabat”、“IMGT”、“Chothia”、“North”或“None”(与 scheme 相同)。当
scheme = AHo时,此参数为必填项。
- 评分相关参数
- 评分助手(Scorer):根据抗体中的各种性质(如人源性、酸碱性和疏水性)对每个氨基酸进行评分的 AI。可选模型包括:
- GeoHumanness:领先的人源性预测模型。
- 侧链pKa:基于结构的侧链 pKa 预测模型。使用此模型时,“Relax structure(松弛结构)”参数将自动设置为“是”。
- wwHydrophobicity (dG water-membrane):Wimley–White 全残基疏水性评分,用于衡量未折叠链从水到双分子层界面的自由能变。
- wwHydrophobicity (dG water-octanol):Wimley–White 全残基疏水性评分,用于衡量未折叠链进入辛醇(与双分子层界面的疏水核心有关)的自由能变。
- wwHydrophobicity (dG difference):dG water-membrane 和 dG water-octanol 之间的差异。
- 突变建议助手(Advisor):为您改造抗体提供突变建议的 AI。可选模型包括:
- ESM1v-650M:擅长预测突变效应的语言模型。具备领先的零样本预测能力。
- ESM2-3B:业界领先的通用蛋白质语言模型。
- GeoHumAb:百奥几何自研的、领先的人源化算法。
- 评分助手(Scorer):根据抗体中的各种性质(如人源性、酸碱性和疏水性)对每个氨基酸进行评分的 AI。可选模型包括:
- 突变相关参数
- Mutator(自动突变助手):将输入抗体序列突变为初始输出的 AI。可选模型如下。您可以前往人源化页面查看这些模型的详细参数。
- None:不进行自动突变。输入抗体将作为初始输出。
- GeoHumAb:领先的人源化算法。
- CDR Grafting:将输入抗体的 CDR 区域嫁接到人源种系上的传统人源化技术。
- Mutator(自动突变助手):将输入抗体序列突变为初始输出的 AI。可选模型如下。您可以前往人源化页面查看这些模型的详细参数。
- 结构预测相关参数
- Relax structure(松弛结构):是否使用 Amber 松弛模型生成抗体的结构。默认为 No(否)。如果使用侧链 pKa 评分助手,则必须为 Yes(是)。
结果面板¶
在文件与任务管理器面板中点击任务结果,可以查看任务结果。 这是您与 AI 助手进行交互式抗体优化的地方。
输入抗体序列显示在标有“Parental (wt)”的行中。 如果指定了自动突变助手,初始的输出抗体将由自动突变助手生成。否则,它将与输入抗体序列相同。 当前的输出抗体序列显示在标有“Current (mt)”的行中。
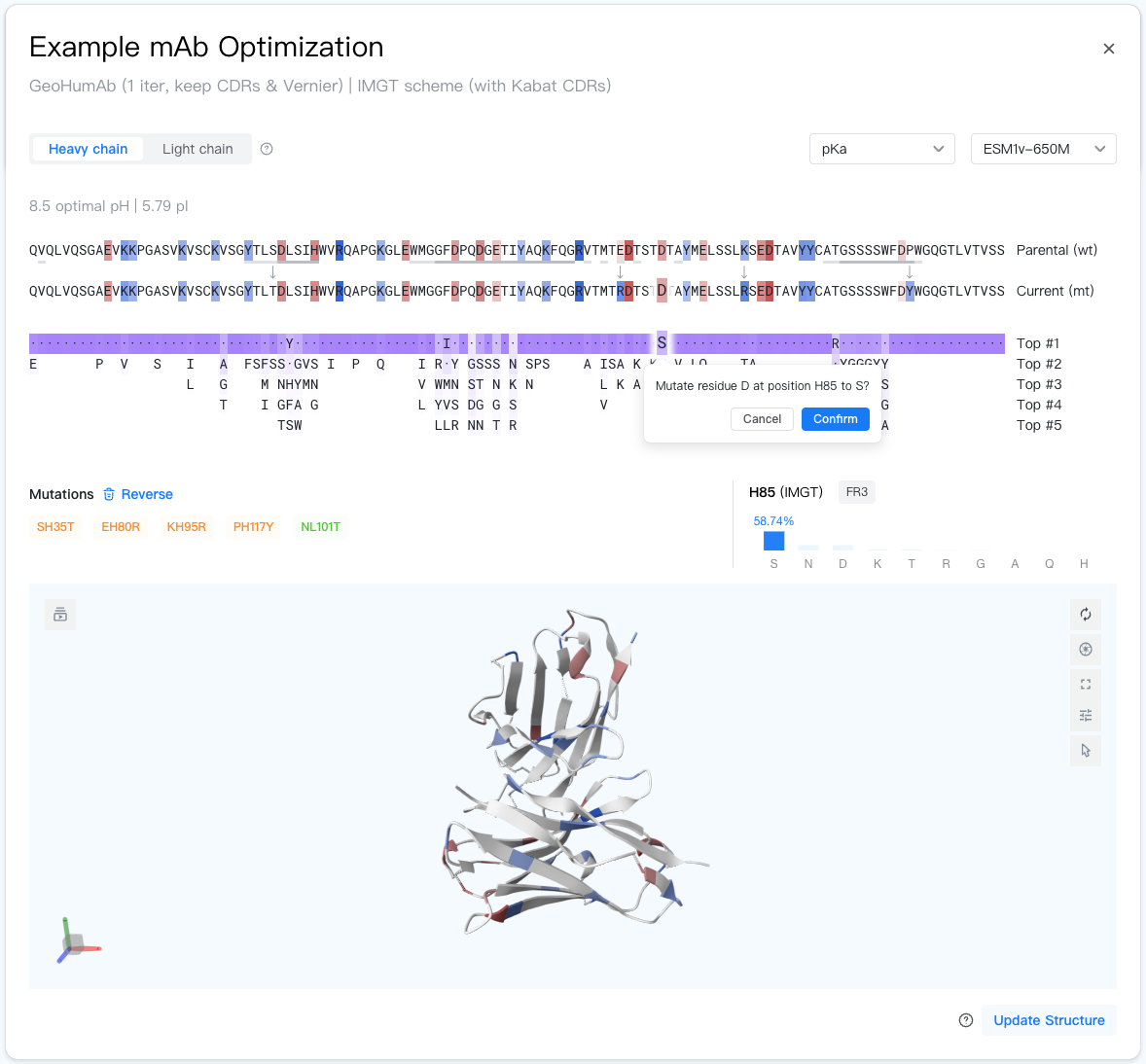
任务小结¶
任务名称和小结显示在结果面板的顶部,作为标题和副标题。 任务小结显示了在此任务中使用的自动突变助手模型和编号方案/CDR 定义。这些参数在提交任务后无法更改。
序列查看器¶
任务小结的下方是序列查看器。您可以在此查看原始和当前的抗体序列,并进行交互式优化。
在查看器左上角,您可以使用链切换器 "" 在两个抗体链之间切换。
在查看器右上角,您可以更改评分助手和突变指导助手使用的模型。
模型和参数部分介绍了有关这些模型的详细信息。
查看每个氨基酸的评分¶
在序列查看器中部,您可以查看原始和当前的抗体序列。每个氨基酸残基的背景颜色展示了评分助手对其的评分。原始和当前序列不同的位置用箭头进行了标记。
当评分助手是 GeoHumanness 时,当前序列中种系频率低于 1% 的氨基酸以红色三角形标记。如果原始序列中某个氨基酸种系频率低于 1%,但当前序列中同一位置的氨基酸种系频率高于 1%,则以绿色三角形标记。
CDR 区域的氨基酸残基以深灰色下划线进行了标记。如果您使用 Kabat 进行 CDR 定义,则 Vernier 区域的氨基酸残基会以浅灰色下划线进行标记。
评分助手的颜色方案是什么?
-
GeoHumanness:白色-红色。
红色越深,免疫原性风险越高。
具体地说,如果一个 9-肽片段在 OAS 数据库人源实验中出现的频率低于 10%,则该片段被认为是高风险肽段。氨基酸属于越多高风险肽段,其红色越深。
-
侧链pKa:蓝色-白色-红色。
红色越深,pKa 越低(越酸性)。蓝色越深,pKa 越高(越碱性)。
-
wwHydrophobicity:青色-白色-黄色。
黄色越深,疏水性越高。青色越深,疏水性越低。
AI 指导的抗体改造¶
在原始和当前抗体序列的下方,您可以查看突变指导助手为您提供的突变建议。 突变指导助手为每个氨基酸提供了置信度最高的 5 个突变建议(当前氨基酸也会参与排名)。置信度越高,突变的背景颜色(紫色)越深。
为了使突变建议更清晰、易读,突变建议中当前序列中的氨基酸残基以点号("·")表示,置信度低于 1% 的突变建议将被省略。
当您将鼠标悬停在突变建议上时,突变建议的详细信息将显示在序列查看器右下角。包括:
- Position(位置):您悬停的氨基酸所在位置的编号。格式为“{链类型}{残基编号}{插入代码}”,例如“H100A”。
- Region(区域):该位置所属的抗体区域,值可能为 FR1-4 或 CDR1-3。如果编号方案设置为 Kabat,则还会标出 Vernier 区域。
- Advisor confidence(突变建议的置信度):排名前 10 的突变建议及其置信度,鼠标悬停处的氨基酸残基以蓝色高亮显示。置信度越高,代表该突变越有可能改善抗体。
如果您想采纳某个突变建议,只需点击该突变建议,随后在弹出窗口中点击“Confirm”按钮即可。 如您想对序列进行自定义突变,您可以点击序列查看器中“Current (mt)”序列中您想突变的氨基酸残基,在弹出窗口中选择一个突变,随后点击“Confirm”按钮。
管理序列突变¶
您可以在”Mutations“区域中查看您已经应用的突变。点击”Reverse“按钮可以选择需要撤销的突变。
结构查看器¶
结构查看器显示了当前抗体的结构。您可以在此查看抗体的结构,并进行交互式优化。 当本任务的结果面板打开时,结构对象面板将自动同步于此面板中的结构。
结构中的氨基酸残基会按照与序列查看器中相同的方案进行着色,以反映评分助手对每个氨基酸的评分。
由于结构预测的推理成本相对较高,因此只有在您点击“Update Structure”按钮或者评分助手设置为“侧链pKa”时,结构才会被更新。 请注意,即使结构对应较老的序列,氨基酸残基的着色始终与当前最新的抗体序列同步。