人源化¶
抗体人源化是将抗体序列改造成更接近人源抗体,从而降低其在人体内的免疫原性的过程
功能亮点¶
- 前沿算法:基于抗体大语言模型的 GeoHumAb 在提升人源性和序列保守性之间取得了很好的平衡。从精度结果可以看出,其预测结果与抗体专家的人源化结果高度一致。
- 直观界面:主要结果清晰易读,种系序列可直观对比,风险位点一目了然。
- 批量预测:支持一个任务提交多条序列到云端批量人源化,效率更高、成本更低。
输入¶
要提交一个人源化任务,请打开项目编辑器,并从“Optimization”(优化)下拉菜单中选择“Humanization”(人源化)。
-
Antibodies(抗体):一个用 FASTA 格式表示的抗体序列的列表。您可以在输入框中输入序列,也可以上传 FASTA 文件 (点击 "
" 按钮)。 每个抗体将独立进行人源化。
- Chain type(链类型):您可以在 Antibodies 标题的右侧选择抗体的链类型,选项有是“VH”、“VL”或“VH+VL”(默认)。如果您想对纳米抗体进行人源化,请选择“VH”。
纳米抗体人源化正在开发中
目前,GeoBiologics 还不支持纳米抗体人源化。我们不会对纳米抗体的框架区域进行反突变(即强制保留原有氨基酸)。如果您使用此模型对纳米抗体进行人源化,请考虑手动反突变这些残基。
- Antibody Name(抗体名称):抗体的名称。默认为“Antibody”。要更改名称,请将鼠标悬停在抗体名称上,然后点击 "
" 按钮。
- Sequence(序列):抗体的序列。每条链的序列长度不得超过 200 个氨基酸。
(上传序列): 点击 "
" 按钮并选择一个 .FASTA 文件上传您自己的抗体。当 Chain type(链类型)为“VH+VL”时,每个抗体必须有两条链,FASTA 标签具有相同的前缀且分别以 "_H" 和 "_L" 结尾。参见以上示例。
- Job Name(任务名称):任务的名称。请注意,任务名称必须在项目内唯一。

模型 & 参数¶
这个任务有两个模型可选:GeoHumAb 和 CDR Grafting。前者是百奥几何自研的抗体人源化模型,后者是将输入抗体的 CDR 区域直接嫁接(graft)到最相近的人源抗体种系上的传统算法。
GeoHumAb 模型的参数如下:
- scheme(编号方案):抗体编号方案。可选“Kabat”、“IMGT”、“Chothia”或“AHo”。
- CDR def.(CDR 定义):用于定义 CDR 区域的编号方案。可选“Kabat”、“IMGT”、“Chothia”、“North”。默认情况下该参数的值与 scheme(编号方案)相同。当 scheme = AHo 时,此参数为必填项。
- niter(迭代次数):GeoHumAb 算法的人源化迭代次数。默认为 1。迭代次数越多,人源化效果越好,但序列保守性越差。
- Keep CDRs(保留 CDR 区域):是否保留 CDR 区域以维持抗体的亲和力。
- Keep Vernier(保留 Vernier 区域):是否保留 Vernier 区域以维持抗体的亲和力。仅当 CDR def. = Kabat 时可用。
CDR Grafting 模型的参数如下:
- scheme(编号方案):抗体编号方案。可选“Kabat”、“IMGT”、“Chothia”或“AHo”。
- CDR def.(CDR 定义):用于定义 CDR 区域的编号方案。可选“Kabat”、“IMGT”、“Chothia”、“North”。默认情况下该参数的值与 scheme(编号方案)相同。当
scheme = AHo时,此参数为必填项。 - VH germline(重链种系):用于人源化的重链种系。默认为“Auto”,即选择与输入抗体最相近的人源重链种系。
- VL germline(轻链种系):用于人源化的轻链种系。默认为“Auto”,即选择与输入抗体最相近的人源轻链种系。
- Keep Vernier(保留 Vernier 区域):是否保留 Vernier 区域以维持抗体的亲和力。仅当
CDR def. = Kabat时可用。
结果¶
结果小结¶
结果小结存储在 CSV 文件中,可以通过点击结果表格右上角的""按钮下载。
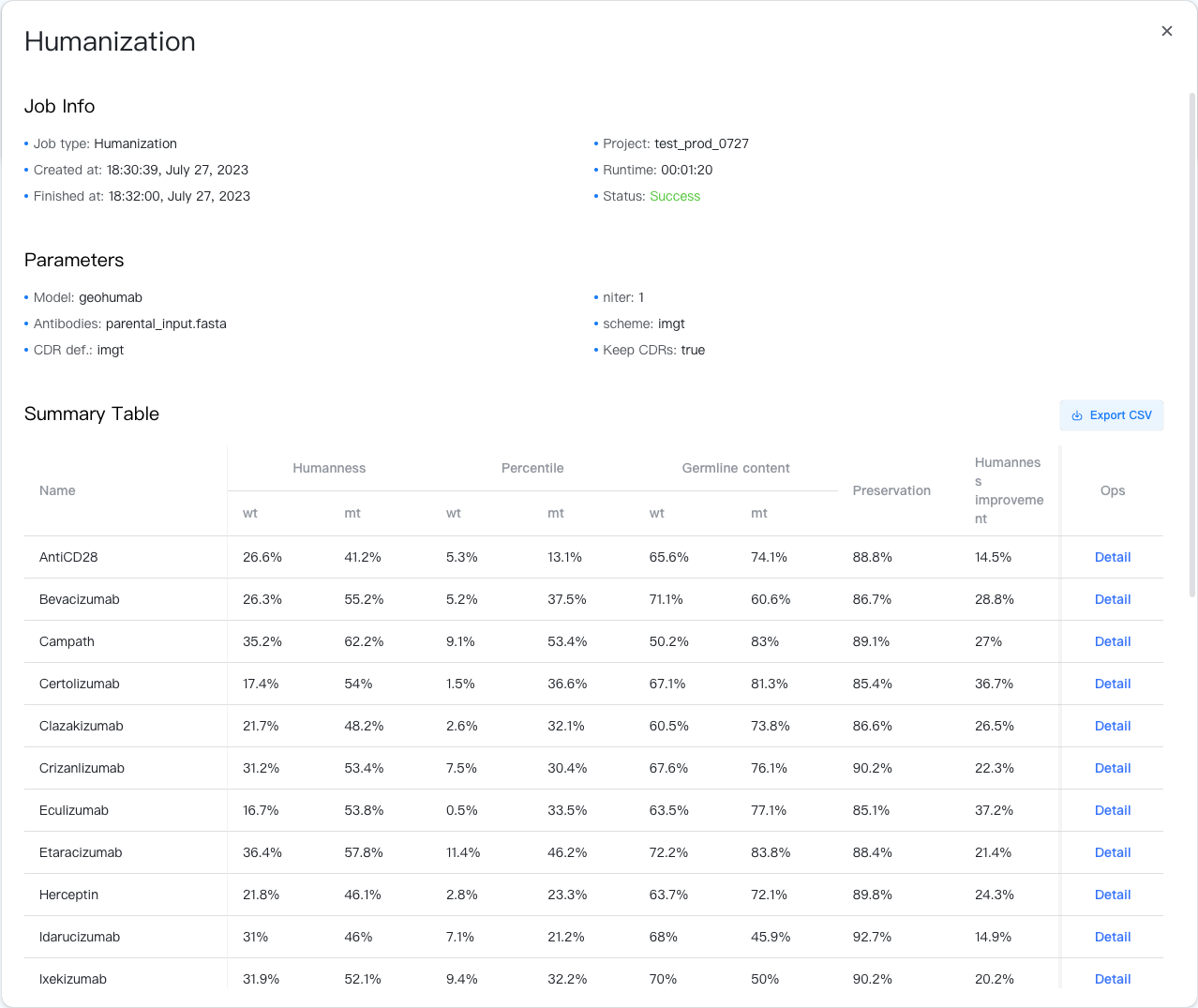
结果表格包含以下列:
- Name(名称):抗体名称,与输入中 FASTA 标签的前缀相同。
- Humanness(人源性):抗体的人源性评分,由抗体中的 9-肽片段在 OAS 数据库人源实验中的平均出现频率决定。得分越高,抗体人源性越好。
- Percentile(百分位):人源性评分在 OAS 数据库中所有抗体中的百分位。得分越高,抗体人源性越好。
- Germline content(种系成分):抗体的种系成分,即抗体与最近的人源轻重链种系序列的序列相似性。
- Preservation(序列保守性):抗体序列与输入抗体序列的相似性。
- Humanness Improvement(人源性提升):人源化前后人源性评分的差值。
在最右侧的列中,您可以点击“Detail”按钮查看某个抗体人源性的详细结果。
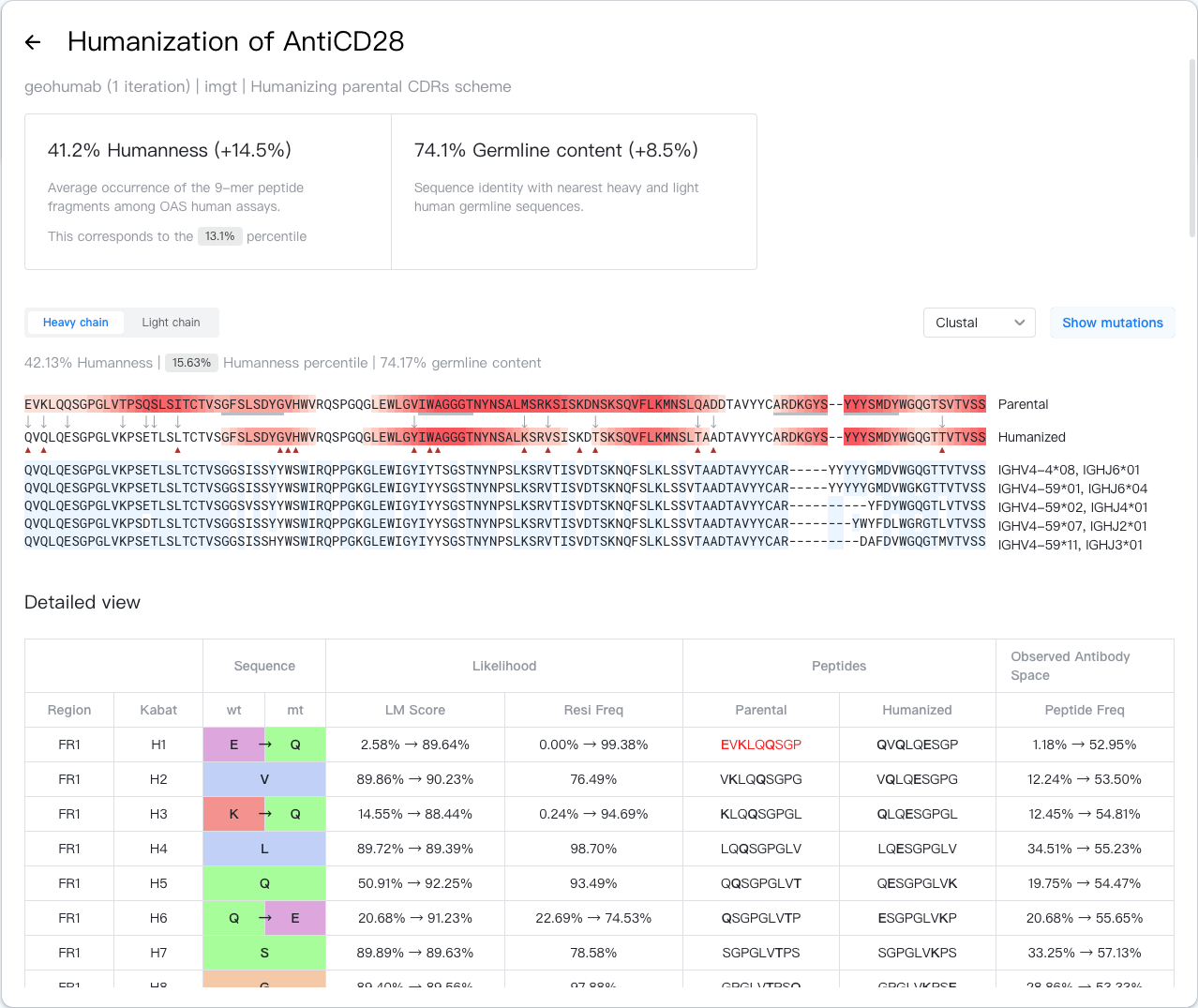
结果详情¶
结果详情页分为 3 个区域:Summary(结果小结)、Germline Comparison(种系比较)和Detailed View(详细视图)。如果输入的链类型为“VH+VL”,种系比较和详细视图区域将分别显示重链和轻链的结果。您可以使用小结部分下方的""链切换器在两条链之间切换。
结果详情的原始数据存储在一个 CSV 文件中。您可以通过点击详情视图区域右上角的""按钮下载。
结果小结¶
结果小结区域显示了任务所用的模型和用于计算人源性的编号方案等设置。它还显示了抗体的人源性评分(及其提升)、百分位和种系成分(如果抗体包含轻重链,显示的值为重链和轻链的平均值)。
序列查看器¶
结果小结区域下方即是序列查看器,其中对齐显示了人源化前后的抗体链。被模型更改氨基酸类型的位置用箭头符号(↓)进行了标记。
序列中种系频率低于 1% 的氨基酸以红色三角形标记,处于高风险肽段中的氨基酸以红色高亮显示。氨基酸属于越多高风险肽段,其红色越深。
CDR 区域的氨基酸残基以深灰色下划线进行了标记。该区域由于 VD(J) 重排和体细胞超突变,可变性较高,因而红色通常较深,但不代表该区域一定存在免疫原性风险。
如果您使用 Kabat 进行 CDR 定义,则 Vernier 区域的氨基酸残基会以浅灰色下划线进行标记。
名词解释
- 种系频率:OAS 数据库相应人源种系中给定位置的氨基酸频率。频率越高,该氨基酸越可能是种系中的氨基酸。
- 高风险肽段:如果一个 9-肽片段在 OAS 数据库人源实验中出现的频率低于 10%,则该片段被认为是高风险肽段。
种系比较¶
种系比较区域对齐显示了人源化前后的抗体链,及与其最接近的五条人源种系序列。种系序列的名称显示在右侧,与目标序列相同的种系残基以蓝色高亮显示。
详情视图¶
详情视图区域以表格形式显示抗体链中每个位置的详细指标。表格包含以下列:
- Region(区域):该位置所属的抗体区域,值可能为 FR1-4 或 CDR1-3。如果编号方案设置为 Kabat,则还会标出 Vernier 区域。
- Pos(位置):按照指定方案编号后的抗体链中的位置编号,格式为“{链类型}{氨基酸编号}{插入码}”,例如“H100A”。
- Sequence(序列):人源化前后,抗体链上该位置的氨基酸类型。氨基酸按照指定的颜色方案进行着色。您可以使用右上角的"
"颜色方案选择器更改颜色方案。
- LM Score(语言模型分数):人源化前后,该位点的语言模型分数。分数越高,语言模型认为该氨基酸在当前上下文中出现的概率最高。
- Resi Freq(种系频率):人源化前后,OAS 数据库相应人源种系中给定位置的氨基酸频率。频率越高,该氨基酸越可能是种系中的氨基酸。
- Peptides(肽段):人源化前后,以该氨基酸为起始氨基酸的 9-肽片段。如果相应的“Peptide Freq”低于 10%,则该片段以红色显示,被认为是高风险肽段。
- Peptide Freq(肽段频率):人源化前后,该 9-肽片在 OAS 人源实验中出现的频率。