酶优化¶
酶优化(Enzyme Optimization)是根据酶序列,优化其包括酶活性,pH 稳定性,热稳定,立体选择性,可溶性等酶学性质的功能。通过分析酶的单点或多点突变对其酶学性质的影响,可以为用户提供定向进化策略,从而实现特定功能的酶改造。
功能亮点¶
酶的催化过程比较复杂,不同酶的催化机理差别较大,且对于不同的底物、相同底物的不同手性具有高度的筛选性。同时,酶的催化能力受温度和 pH 等环境条件的影响较大,传统方法很难同时考虑反应中的各种条件。对此,GeoBiologics 中的酶优化模块有着如下优势:
-
先进算法: GeoEnzyme 使用最新的几何深度学习技术,处理整个酶促反应与酶的交互作用。特别地,GeoEnzyme 充分考虑酶对小分子反应物与产物的手性选择性,可以提供更加精确的预测。
-
支持指定反应条件: GeoEnzyme 考虑了反应体系中不同温度和 pH 对酶催化过程的影响。用户可以指定反应体系中的温度和 pH 等反应条件,进行更加符合实际应用场景的模拟。
-
易用: GeoEnzyme 支持同时对指定的单点和多点突变进行筛选。您可以很清晰地比较不同突变策略带来的酶学性质影响,迭代酶的进化方向。
输入¶
要提交酶优化任务,请打开项目编辑器并点击左侧边栏中的"New Job(新建任务)"按钮,然后选择"Protein Design(蛋白设计)"任务组中的"Enzyme Optimization(酶优化)"。任务提交表单将在新标签页中打开。

-
Enzyme Sequence(酶序列): 待优化的酶序列,输入序列长度不能超过 1000 个氨基酸。
-
Reactants(反应物): 酶促反应中的反应物,输入为 SMILES 表达式。输入框中,每行输入一个 SMILES,支持输入手性分子。输入方式详见使用 Ketcher。
-
Products(产物): 酶促反应中的产物,输入格式与Reactants相同。
-
By-reactants(副反应物): 当 Objective 选择 “Stereoselectivity” 时,用于表示在酶促反应中的副反应物,输入格式与Reactants相同。副反应物与反应物之间除手性外应完全相同;如果反应物不带手性,或手性不影响反应,则副反应物与反应物应完全一致。
-
By-Products(副产物): 当 Objective 选择 “Stereoselectivity” 时,用于表示在酶促反应中的副产物,输入格式与Reactants相同。副产物与产物之间仅手性构型不同。当产物中有多个手性中心时,请填写主要副产物对应的手性异构体。
-
Mut. mode(突变模式): 突变模式分为 “Saturation Mutagenesis” 和 “Custom” 两种。“Saturation Mutagenesis” 表示单点饱和突变,“Custom” 表示自定义单点或多点组合突变。
-
Mutation sites(突变位点): 当突变模式为 “Saturation Mutagenesis” 时,需要输入突变位点。输入使用逗号分隔,例如:32-36 或者 100,102-105,其中 32-36 表示对应闭区间,即位点 32,33,34,35,36。
-
Mutation list(突变列表): 当突变模式为 “Custom” 时,需要输入突变列表。每行表示一个单点或多点突变,多点突变内部使用逗号分隔,例如:G25T(单点突变)或者 A35W,Q64F,E103T(三点突变)。
-
-
Job Name(任务名称): 任务的名称。请注意,任务名称必须在项目内唯一。
模型 & 参数¶
您可以使用我们自研的 GeoEnzyme 模型或者酶优化(训练)中自定义训练的模型(模型名称与训练任务名称相同)来运行本任务。模型的参数如下:
-
Objective(优化目标): 您可以在 “Enzyme Activity” “pH Stability” “Thermostability” 和 “Stereoselectivity” 之间选择一个或多个优化目标。
-
“Enzyme Activity” 表示优化酶的活性;
-
“pH Stability” 和 “Thermostability” 分别表示优化酶的 pH 稳定性和热稳定性,通过指定具体的酶促反应体系条件,来优化酶在该指定条件下的催化能力。
-
“Stereoselectivity” 表示优化酶的立体选择性,其中,产物与主要副产物的手性需要在输入中指定;
-
“Solubility” 表示优化酶的可溶性,默认表达体系为大肠杆菌(Escherichia coli)。
-
-
pH(酸碱度): 当目标选择了 “pH Stability” 之后,可以指定目标体系中的 pH 值,取值范围为 0-14,默认值为 7。
-
Temperature(温度): 当目标选择了 “Thermostability” 之后,可以指定目标体系中的温度,取值范围为 0-100,默认值为 37。
结果¶
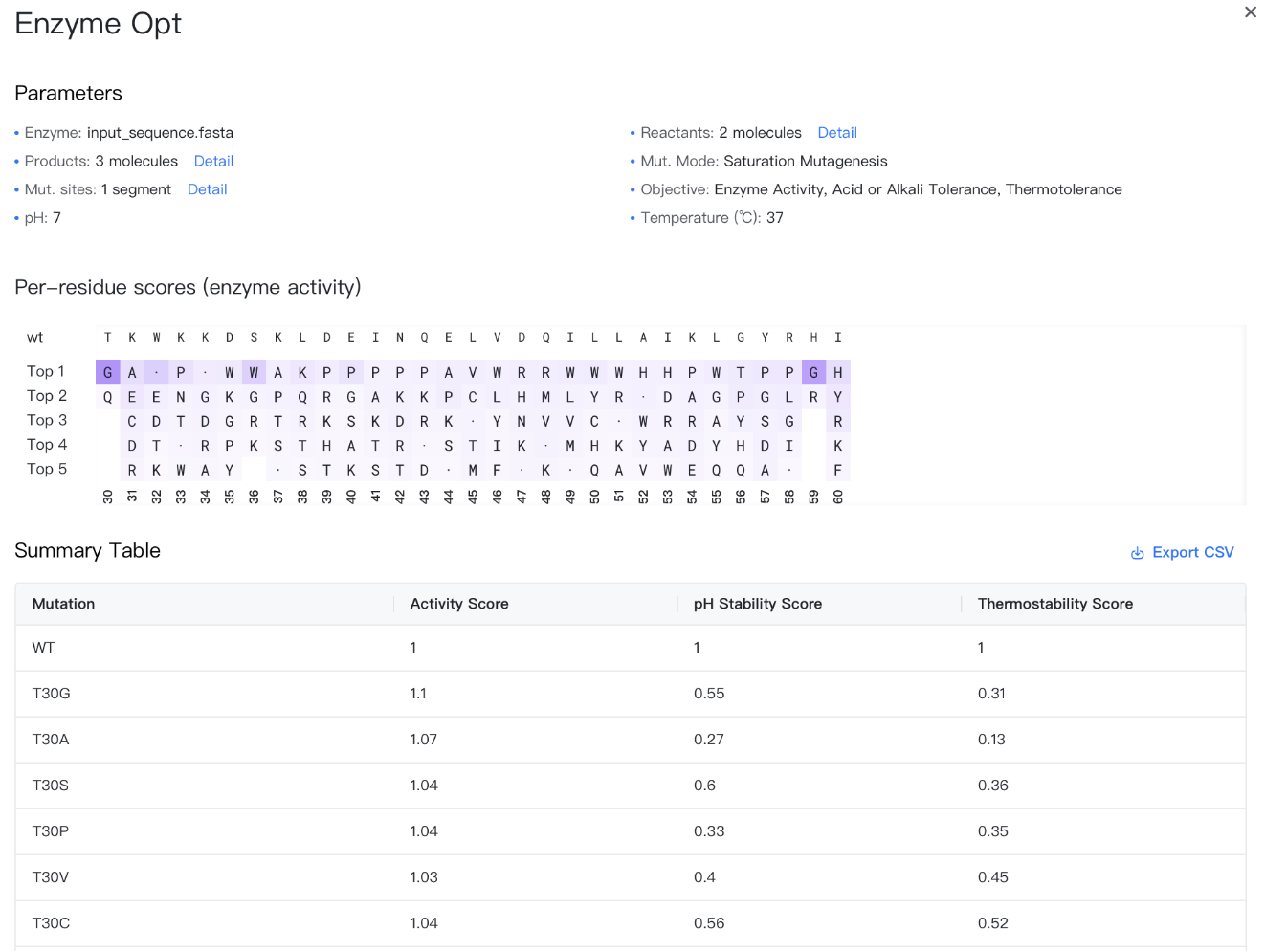
运算结果¶
结果存储在 CSV 文件中,可以通过点击结果表格右上角的""按钮下载。
结果表格包含以下列:
-
Mutation(突变): 与输入相同的点突变标签,格式为“原氨基酸+残基ID+突变氨基酸”,例如
F103Y。表格第一行固定为“WT”,表示突变前的野生型酶。 -
Activity Score(酶活性分数): 突变导致的酶活性变化,大于 0 表示酶活提高,小于 0 表示酶活降低,等于 0 表示酶活基本不变。第一行“WT”对应的酶活性分数固定为 0。
-
pH Stability Score(pH稳定性分数): 突变导致的酶 pH 稳定性变化,表示酶在指定 pH 下的活性,大于 0 表示 pH 稳定性提高,小于 0 表示 pH 稳定性降低。第一行“WT”对应的酶pH 稳定性分数固定为 0。
-
Thermostability Score(热稳定性分数): 突变导致的酶热稳定性变化,表示酶在指定温度下的活性,大于 0 表示热稳定性提高,小于 0 表示热稳定性降低。第一行“WT”对应的酶热稳定性分数固定为 0。
-
Stereoselectivity Score(立体选择性分数): 突变导致的酶的立体选择性变化,大于 0 表示立体选择性提高,小于 0 表示立体选择性降低。第一行“WT”对应的酶立体选择性分数固定为 0。
-
Solubility Score(可溶性分数): 突变导致的酶的可溶性变化,大于 0 表示可溶性提高,小于 0 表示可溶性降低。第一行“WT”对应的酶可溶性分数固定为 0。
突变建议¶
如果任务的突变模式选择了饱和突变(saturation mutagenesis),结果页面中还会根据您选择的优化目标(Objective)出现对应的“Per-residue scores”,展示每个突变位点,在该优化方向上最推荐的 5 个氨基酸。
- wt 一行标出了原始分子中突变位点的氨基酸。
- Top 1-5 分别列出了每个位点最推荐的 5 个氨基酸。
- 打分较低的氨基酸不会被展示,自突变除外。
- 每个单元格内会显示 mt_aa,即突变后的氨基酸。自突变的单元格内显示“·”,以方便您快速定位自突变的位置。
- 单元格的背景色展示了当前氨基酸在当前突变位点所有20种可能的氨基酸中所占的优势。如果自突变的背景色很浅,说明该位点提升的潜力较大。
当您将鼠标悬浮在某个单元格上时,会出现一个提示框,展示当前突变体突变前后的氨基酸。