蛋白优化¶
蛋白优化是指优化蛋白的各项性质,包括适应性、热稳定性和溶解性。通过分析蛋白的单点或多点突变对其性质的影响,用户可以制定更合理的定向进化策略,为蛋白改造提速增效。
输入¶
要提交酶优化任务,请打开项目编辑器并点击左侧边栏中的"New Job(新建任务)"按钮,然后选择"Protein Design(蛋白设计)"任务组中的"Protein Optimization(蛋白优化)"。任务提交表单将在新标签页中打开。

-
输入模式(Input Mode):您可以选择以下输入模式之一:
- 基于序列(Sequence-based):将使用 ESMFold 从蛋白序列预测结构。仅在蛋白结构不可用时选择此选项。
- 蛋白序列(Protein Sequence):待优化的蛋白序列,最大长度为 1000 个氨基酸。
- 基于结构(Structure-based):使用蛋白序列和结构来提供更可靠的结果。
- 结构选择器(Structure Selector):从项目或当前查看器中选择要优化的结构。
- 待优化链(Chain to opt.):结构中要优化的链 ID。
- 基于序列(Sequence-based):将使用 ESMFold 从蛋白序列预测结构。仅在蛋白结构不可用时选择此选项。
-
突变模式(Mut. mode):突变模式可以是"饱和突变"或"自定义"。"饱和突变"表示单位点饱和突变,而"自定义"允许自定义单个或多个突变组合。
-
突变位点(Mutation Sites):以逗号分隔的突变位点编号的列表(从 1 开始),例如
32-35,100,102-105,其中32-35表示闭区间,包括位点 32、33、34 和 35。 -
突变列表(Mutation list):突变列表,每行表示一个携带一个或多个突变的突变体,例如
G25T(单突变体)或A35W,Q64F,E103T(三突变体)。多个突变之间用逗号分隔。
-
-
任务名称:任务的名称。请注意,任务名称在项目内必须是唯一的。
模型与参数¶
您可以使用百奥几何自研的 GeoProt 模型来运行此任务。该模型的参数如下:
-
优化目标:您可以从"适应性"、"热稳定性"和"溶解性"中选择一个或多个优化目标
- "适应性"是AI大模型对蛋白生存概率和功能强弱进行拟合得到的通用指标;
- "热稳定性"指蛋白在加热条件下的稳定性。
- "溶解性"指蛋白在水中的溶解度。
结果¶
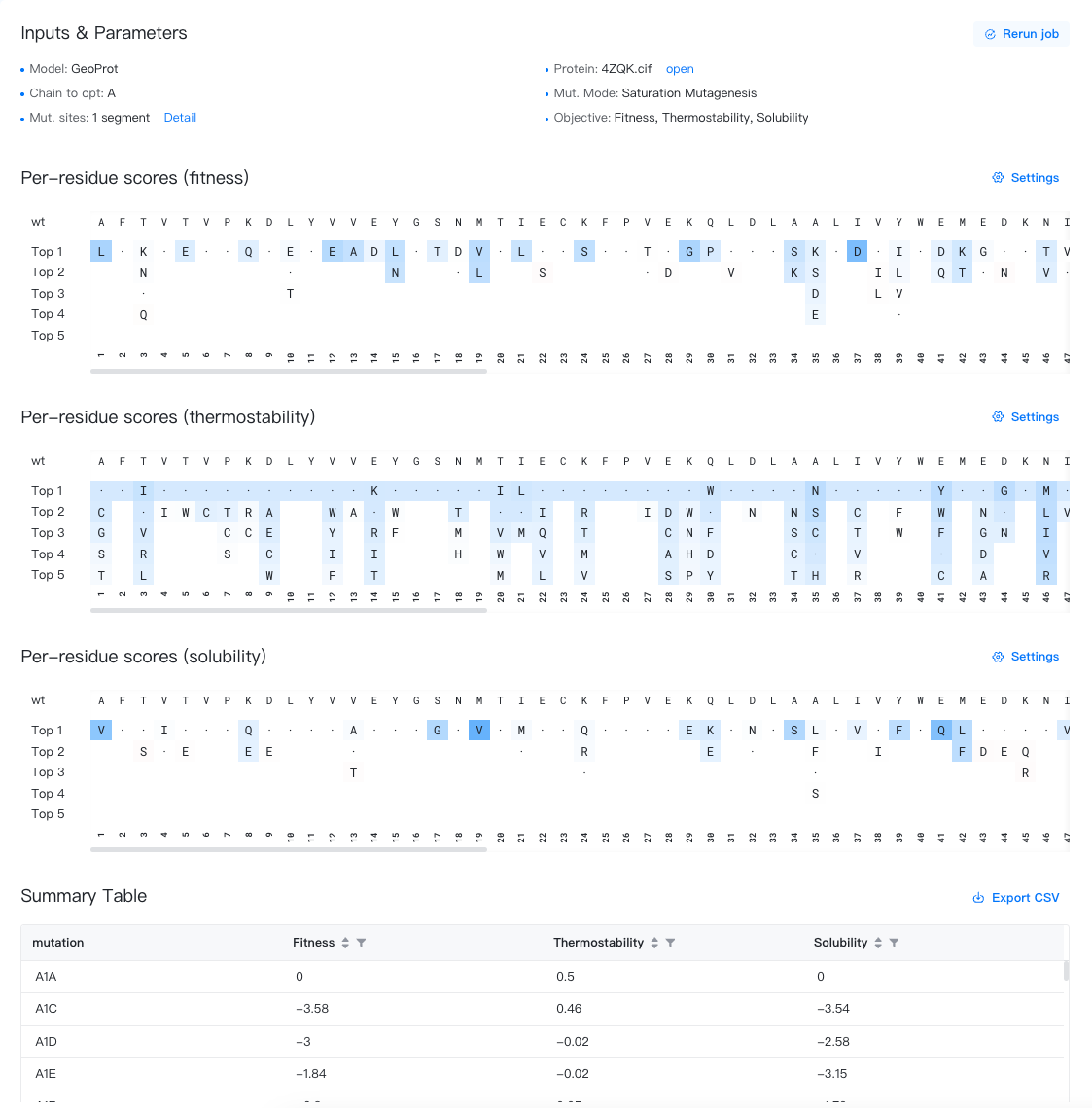
汇总表格¶
结果存储在 CSV 文件中,可以通过点击汇总表右上方的""按钮下载。
汇总表包含以下列:
-
Mutation(突变):点突变的标签,格式为"原始氨基酸 + 残基编号 + 突变氨基酸",例如 F103Y。第一行固定为"WT",代表突变前的野生型酶。
-
Fitness(适应性):由于突变引起的适应性变化。得分大于 0 表示适应性提高;得分小于 0 表示适应性降低。
-
Thermostability(热稳定性):由于突变引起的热稳定性变化。得分大于 0 表示热稳定性提高;得分小于 0 表示热稳定性降低。
-
Solubility(溶解性):由于突变引起的溶解性变化。得分大于 0 表示溶解性提高;得分小于 0 表示溶解性降低。
突变建议¶
如果任务的突变模式选择了饱和突变(saturation mutagenesis),结果页面中还会出现一个“Per-residue scores”区域,展示每个突变位点上最推荐的 5 个氨基酸。
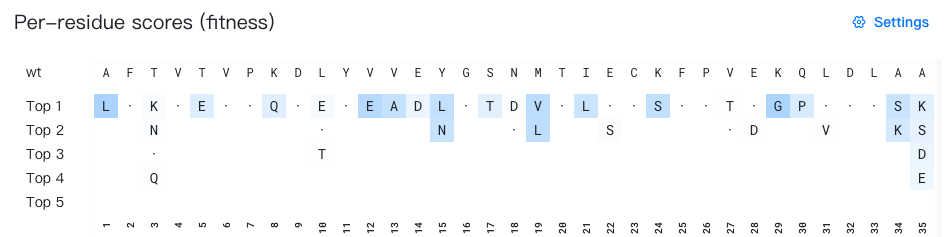
- "wt" 一行标出了原始分子中突变位点的氨基酸。
-
"Top 1-5" 分别列出了每个位点最推荐(亲和力最强)的5个氨基酸。
每个单元格内会显示 mt_aa,即突变后的氨基酸。自突变的单元格内显示“·”,以方便您快速定位自突变的位置。突变概率较低的突变会被隐藏。
单元格的背景色展示了突变成该氨基酸带来的性质变化。
- 在“原始分数”模式(默认模式)下,性质提升的突变会被着色为蓝色,性质降低的突变会被着色为红色。
- 在“位点内概率”模式下,性质提升的突变会被着色为紫色。注意,在本模式下,概率值会按位点分别进行归一化,不同位点的概率值是不可比较的。
当您将鼠标悬浮在某个单元格上时,会出现一个提示框(如“3 | T → K | score = 0.17”),展示当前突变体突变前后的氨基酸,以及性质变化的预测值和 95% 置信区间。