序列设计¶
序列设计是生成与给定蛋白质/抗体结构相兼容的序列的任务。
本任务首先基于目标结构生成序列,然后使用 AF2-Multimer 预测设计序列的结构,通过一系列虚拟筛选指标,优选出与目标结构一致性最高的序列。
功能亮点¶
- 经验证的算法:支持已被实验验证、受到广泛使用的蛋白序列设计算法 ProteinMPNN。经微调后可以用抗体序列设计。
- 定制化设计:支持自定义氨基酸分布和可溶性偏好,让设计出的序列更符合您需求。
- 云端批量设计:该算法部署在我们的云平台上,并可针对一个结构批量设计多条序列,进而能够大规模运行。算法设计的结果以 csv 格式储存,方便导出。
输入¶
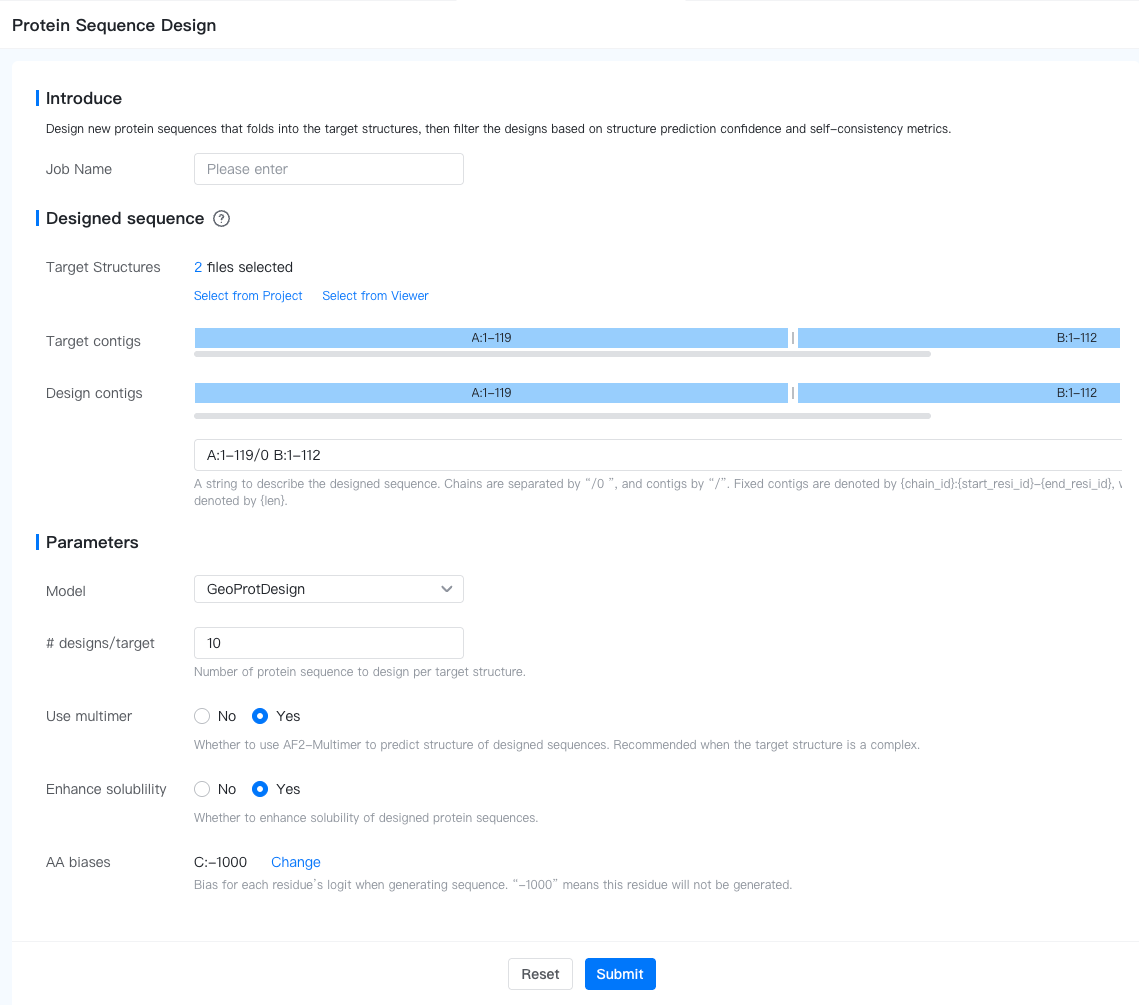
要提交序列设计任务,请打开项目编辑器,并点击左侧边栏中的"New Job(新建任务)"按钮,然后选择"Protein Design(蛋白设计)"任务组中的"Protein Sequence Design(序列设计)"。任务提交表单将在新标签页中打开。
- Job Name(任务名称):任务的名称。请注意,任务名称必须在项目内唯一。
- Designed sequence(要设计的序列):这里我们指定要设计的目标结构中的序列片段。
- Target Structures(目标结构):要设计序列的目标结构,以 PDB/mmCIF 格式表示。结构可以从本地机器上传,从云数据库导入或通过 GeoBiologics 任务生成。
- Target Contigs(目标片段):这是从目标结构中自动生成的非可编辑字段。它包含目标结构的片段描述(如输入多个目标结构,它们必须具有相同的contigs,否则平台将报错)。
- Design Contigs(要设计的片段):这里我们指定目标结构中要设计的的和要固定的序列片段。
- Fixed contig(固定片段):该片段的序列将复制自目标结构。表示为
{chain_id}:{start_resi_id}-{end_resi_id}或{chain_id}:{resi_id}。 - Designed contig(要设计的片段):该片段的序列将从头设计。表示为
{min_len}-{max_len}或{len}。
- Fixed contig(固定片段):该片段的序列将复制自目标结构。表示为
模型 & 参数¶
GeoProtDesign 是基于 ProteinMPNN 的蛋白序列设计模型,已在多个蛋白设计任务中得到验证。
该模型的参数如下:
- # designs/target (生成的设计数量): 每个目标结构要生成的序列数量。默认值为 10。
- Use multimer (是否使用 AF2-Multimer): 是否使用 AF2-Multimer 预测设计序列的结构。推荐在目标结构为复合物时使用。
- Enhance solubility (是否增强可溶性): 是否增强设计序列的可溶性。
- AA biases (氨基酸偏好): 生成序列时每个氨基酸的偏好。"-1000" 表示该氨基酸不会被生成。
结果¶
在文件与任务管理器面板中点击任务名称,查看任务结果。
任务小结¶
任务小结展示了设计后序列文库、虚拟筛选条件、筛选后序列文库的基本信息。
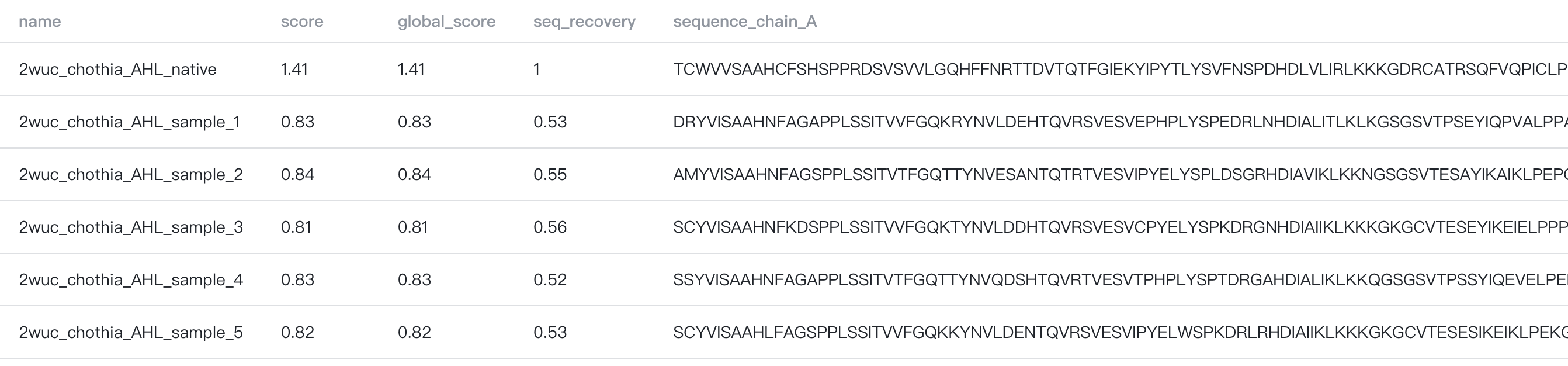
- Designed sequences(设计后的序列文库): 粗体数字显示了设计后序列文库的大小。
: 点击查看前 500 个设计的序列及其打分。
: 鼠标悬浮后会弹出悬浮菜单,您可点击下载(1)AI 模型设计的序列及其打分;(2)设计出的完整抗体序列;(3)所有设计的复合物结构。
- Logo 图:下方的 logo 图绘为您展示设计出的所有序列的氨基酸分布。
- Filtered sequences(筛选后的序列文库): 粗体数字显示了筛选后序列文库的大小。
: 点击查看前 500 个筛选后的序列及其打分。
: 鼠标悬浮后会弹出悬浮菜单,您可点击下载(1)AI 模型筛选后的序列及其打分;(2)完整筛选的序列。
- Filtering criteria(筛选条件): 虚拟筛选的筛选条件。每个条件旁边会用灰色字显示通过筛选的序列所占的比例。您可以点击“Edit”来更改筛选条件,筛选后的序列文库会随之更新。
虚拟筛选中使用的打分项
虚拟筛选环节,我们会使用自研的 GeoFlow 模型评估设计出来的复合物结构与序列之间的一致性(consistency),或者说匹配度。具体打分指标包括:
- mpnn: 序列设计负对数似然(negative log-likelihood)。分数越小,表示序列越可能被模型采样到。
- plDDT: 预测的复合物所有残基的 lDDT (local Distance Difference Test) 分数的平均值。分数越高,表示复合物整体结构一致性越高。
- pTM: 预测的 TM (Template modeling) 分数。分数越高,表示复合物结构的整体置信度越高。注意,这个分数是整个复合物的平均值,所以容易被某个大的结构域主导。
- ipTM: 预测的复合物接口区(antibody-antigen interface)的 TM 分数。分数越高,表示复合物结合界面的置信度越高。对于单体蛋白,这个分数总是 0。
- pAE: 预测的复合物所有残基的 aligned error (AE) 的平均值。分数越低,表示复合物整体序列-结构一致性越高。
- RMSD: 预测与设计的复合物结构之间,抗体的均方根偏差(RMSD)。分数越低,表示蛋白序列-结构一致性越高。
打分的统计量¶
本节用表格展示了各打分指标在所有设计序列上的统计量,包括均值、标准差、最小值、最大值,以及 5%、10%、25%、50%、75%、90% 和 95% 百分位数。您可以基于这些统计量来理解打分的分布,并设置筛选条件。
