抗体-抗原对接¶
抗体-抗原对接(Antibody-Antigen Docking)是根据抗原和抗体的序列来预测抗体-抗原复合物结构的功能,帮助用户了解抗体-抗原以何种方式结合、结合位点的位置等信息。
实际应用中,由于算法对结构的预测存在误差,我们会返回多个预测结果,并附带每个预测结构的质控指标供您参考。
功能亮点¶
蛋白系统的巨大尺寸使得确定两个蛋白的最佳排列变得非常困难。而且,在对接过程中,两个蛋白的灵活区域通常会发生构象变化。GeoBiologics 中的抗体-抗原对接模块具有如下优势:
-
先进算法: 通过利用最新的几何深度学习技术和大规模结构预训练,GeoFlow V2 在抗体-抗原对接方面展现出强大且稳健的性能,成功率比 AlphaFold3 复现模型提高了 ~30%,比 AlphaFold2 提高了 ~93%。
-
灵活的约束支持:您可以输入抗原的表位区域和部分/全部的抗原结构,来引导对接结果。在提供4个表位氨基酸的情况下,对接成功率可以提升从 ~45% 大幅提升至 ~75%。
-
一站式分析:我们深知您面对大量预测结构时分析、对比的痛苦。平台会自动为您对齐所有预测结构的抗原部分并进行表位聚类,让您更清晰地了解预测结构的表位分布。同时,所有预测结构都附带一系列质控指标,帮助您评估预测结果的可靠性。当您打开一个预测结构时,界面可视化面板会自动弹出,列出所有抗体-抗原相互作用,方便您进行分析。
输入¶
要提交抗体-抗原对接任务,请打开项目编辑器并点击左侧边栏中的"New Job(新建任务)"按钮,然后选择"Structure Modeling(结构建模)"任务组中的"Antibody-Antigen Docking(抗体-抗原对接)"。任务提交表单将在新标签页中打开。
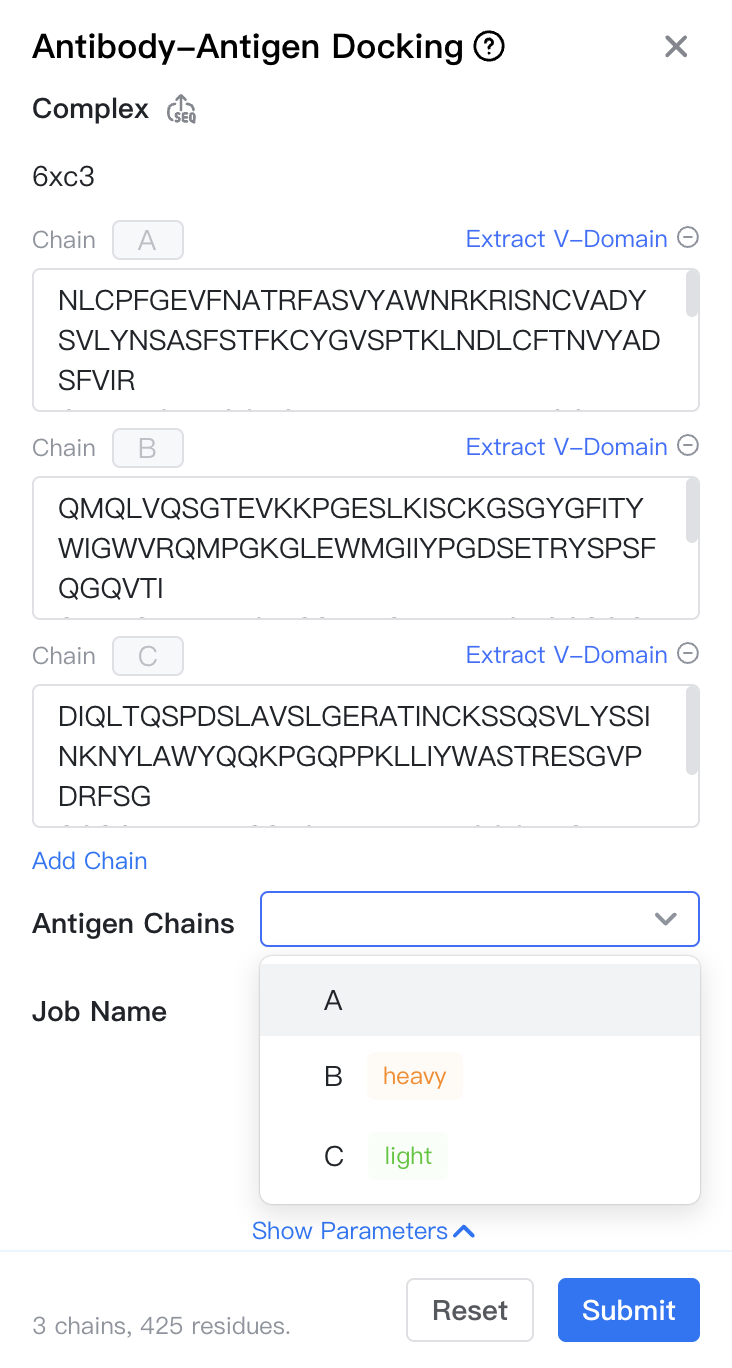
-
Complex(复合物): 抗原-抗体复合物序列。序列有多种输入方式
-
点击 "
" 按钮上传您自己的抗原-抗体复合物(一个 .FASTA 文件)。文件中只能存在一个抗原-抗体复合物,各链的 FASTA 标签必须具有相同的前缀且以
_{chain_id}等结尾。 -
直接在序列输入框中复制 FASTA 字符串内容,平台会自动解析并创建多链序列。FASTA 字符串同样需要满足上述要求。您可以将以下序列复制到序列输入框中以测试此功能:
>6xc3_C NLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIR GDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVNYNYLYRLFRKSNLKPFERDISTEIYQA GSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPK >6xc3_H QMQLVQSGTEVKKPGESLKISCKGSGYGFITYWIGWVRQMPGKGLEWMGIIYPGDSETRYSPSFQGQVTI SADKSINTAYLQWSSLKASDTAIYYCAGGSGISTPMDVWGQGTTVTVSS >6xc3_L DIQLTQSPDSLAVSLGERATINCKSSQSVLYSSINKNYLAWYQQKPGQPPKLLIYWASTRESGVPDRFSG SGSGTDFTLTISSLQAEDVAVYYCQQYYSTPYTFGQGTKVEIK- 在序列输入框中一次输入每条链的序列。注意链的标号固定为 ABCDEF。
-
-
Antigen Chains(抗原链): 选择哪些链属于抗原,剩下的链默认属于抗体。
链类型识别
GeoBiologics 会自动识别并标出结构中的 V-结构域,包括抗体重链(heavy)、抗体轻链(light)、TCR 重链(alpha)、TCR 轻链(beta)。
-
Provide Structure(提供结构): 如您已知抗原的完整/部分结构,可以点击抗原链 ID 下方的 "provide structure" 按钮,在弹出的浮窗中为抗原提供结构。
- Reference Structure(参考结构): 选择参考结构的来源。
- Autofill from structure selection(从结构选区自动填充): 点击此按钮,将选中的序列复制到 "Input Sequence(输入序列)" 中,然后根据结构中存在的片段自动填充结构-序列关系表。
- Input Sequence(输入序列): 抗原的序列。此输入框与外部的序列输入框同步。
- Sequence-Structure Relation Table(结构/序列片段): 在每一行中,输入参考结构中某一连续片段的 label_seq_id 与相匹配的序列 ID(从1开始),以完成序列-结构的匹配。您可以通过如下操作获取 label_seq_id: (1) 选择参考结构中的片段,(2) 将鼠标悬停在
按钮上,(3) 点击 "Copy selected sites (label)"。

任务提交表单:提供结构 警告
- 如果您为多条链提供结构,所有您提供的结构之间的相对位置关系会被保持,就好像它们来自同一个复合物结构一样。在绝大多数情况下,您无需为抗体提供结构。
- 您可以只提供结构中的α-螺旋/β-折叠区域来在对接过程中对回环(loop)区域进行建模。
-
Constraints(约束): 您可在此指定抗原上的表位氨基酸(通过链 ID 和序列 ID 定位),即与特定抗体链结合的氨基酸。
-
Job Name(任务名称): 任务的名称。请注意,任务名称必须在项目内唯一。
模型 & 参数¶
您可以使用我们自研的 GeoFlow V3 模型来运行本任务。此模型的参数如下:
- Mode(模式): 您可以在 “fast(快速)“ 和 ”accurate(精确)“ 之间选择,前者速度更快,后者会进行更多对接姿态的采样,对接成功率更高。
- Relax structure(松弛结构): 如果设置为 "Yes",AI 生成的结构会经历进一步松弛(relax),以优化结合界面氨基酸侧链的构象。请注意,如果输入中存在非标准残基,此选项只能设置为 "No"。
结果¶
结构聚类¶
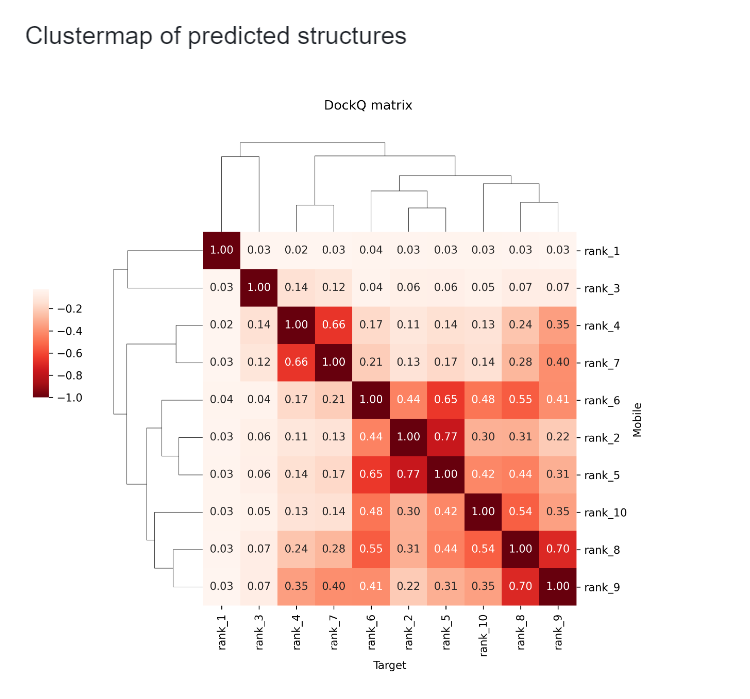
"Clustermap of predicted structures(预测结构聚类图)"这一节展示了所有预测结构的聚类结果。 每个方块内标注了两个结构之间的 ligRMSD 值,即在抗原对齐后两个抗体结构的 RMSD 值。 通过这张图,您可以轻松了解哪几个结构是相似的,哪几个结构是不同的。从而减少查看结构的工作量。您也可以通过 "cluster_label" 列来查看每个结构所属的聚类。
汇总表格¶
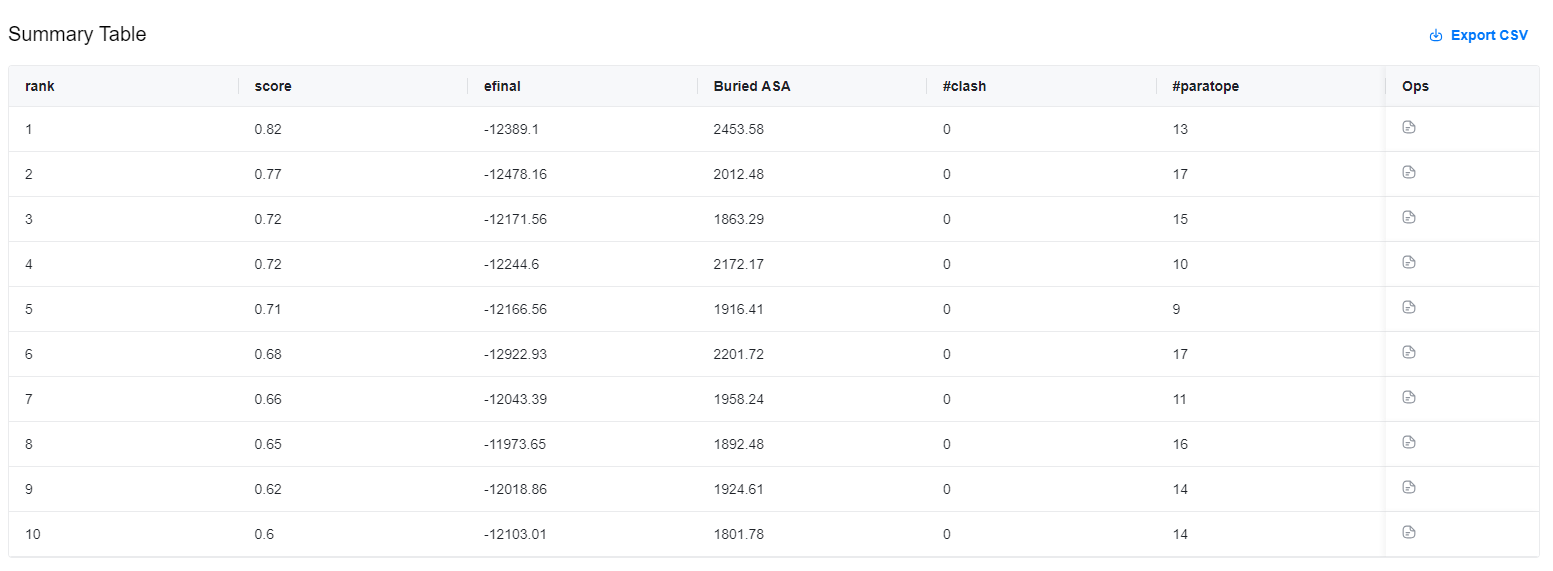
汇总表格中展示了预测的10个结构的置信度和质控指标,具体包括:
- name(名称): 预测结构的名称,其中包含从 0 开始的排名。
- rank_by(综合评分):用于排序的单一指标。数值越小越好。
- dG_bind(结合自由能变):抗体与抗原的结合自由能变。数值越小越好。仅在 "Relax structure" 设置为 "Yes" 时显示。
- cluster_label(聚类标签):预测结构所属的聚类标签。同一聚类中的结构,其 ligRMSD 值通常小于 8 Å。
- ipTM(抗原抗体界面TM-score):抗原抗体界面TM-score,数值越大越好。
上述汇总表可以通过点击表格右上角的""按钮下载。
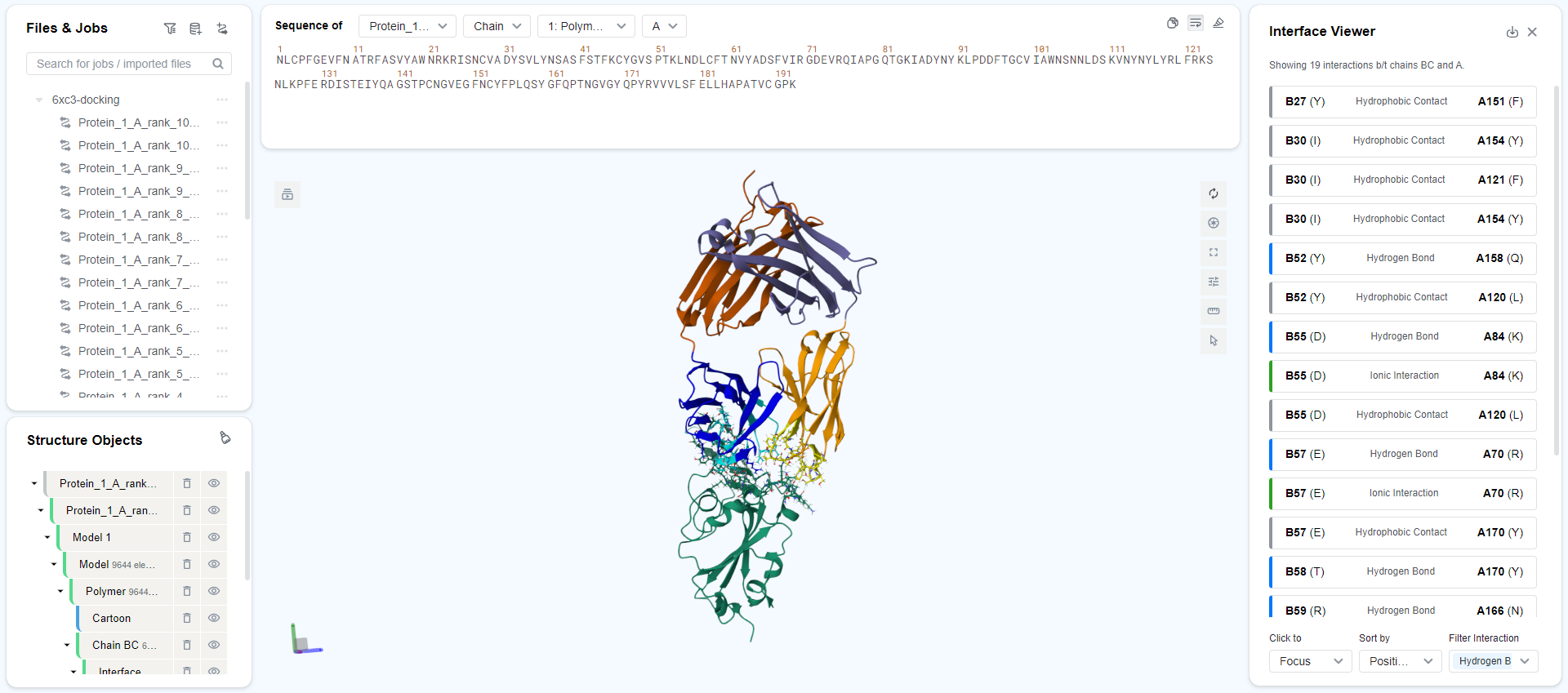
右侧的操作栏(Ops)中,您可以点击 "" 按钮查看复合物结构。点击后将自动跳转到 Mol* 查看器,并打开界面可视化工具,让您查看预测的复合物结构与界面情况。
所有预测的结构中的抗原已经提前为您对齐,您可以打开多个结构比较它们抗体位置的差异。如您需要对齐预测和真实的复合物结构,请参见结构对齐任务文档。
相关链接¶
基于预测的抗原-抗体复合物结构,您可以执行基于结构的亲和力优化,以提高/降低您的抗体-抗原复合物的亲和力。